在 Terra 上运行 NVIDIA Parabricks
本指南展示了如何在 Terra 上运行 Parabricks。
Parabricks 是一个加速计算框架,支持基因组学行业的各种应用,主要支持 DNA、RNA 和体细胞突变检测应用的分析工作流程。凭借行业领先的计算时间,Parabricks 可以快速将 FASTQ 文件转换为 VCF,使用多种经过行业验证的变异调用器,并且还能够对这些变异进行 QC 和注释。由于 Parabricks 基于公开可用的工具,因此结果易于验证,并且可以与其他公开可用的数据集结合使用。
更多信息请访问 Parabricks 产品页面。
详细的安装、使用和调优信息请参考 Parabricks 用户指南。
在 Terra 上,NVIDIA 团队创建了一个 Parabricks 工作空间,其中包含预加载的数据和工作流程配置。在本教程中,我们将从这里开始。这将带我们进入 Terra 上可用的工作流程目录。
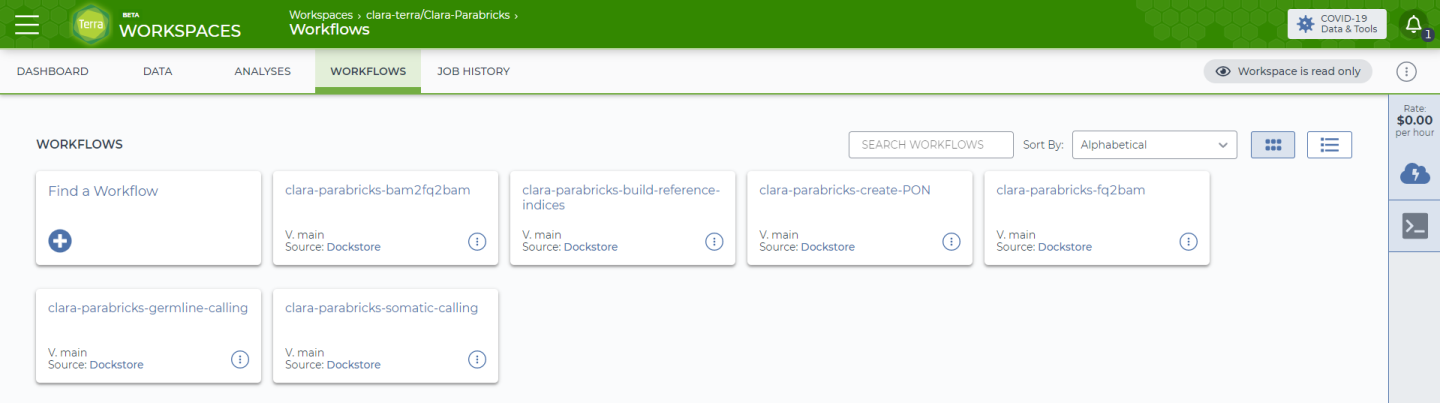
此工作空间为只读,因此要对其进行任何操作,我们必须克隆它。单击右上角的三个点,然后选择“克隆”。为项目命名、选择计费项目和位置,然后选择“克隆工作空间”
我们可以点击返回工作空间并选择“clara-parabricks-fq2bam”。页面应如下所示
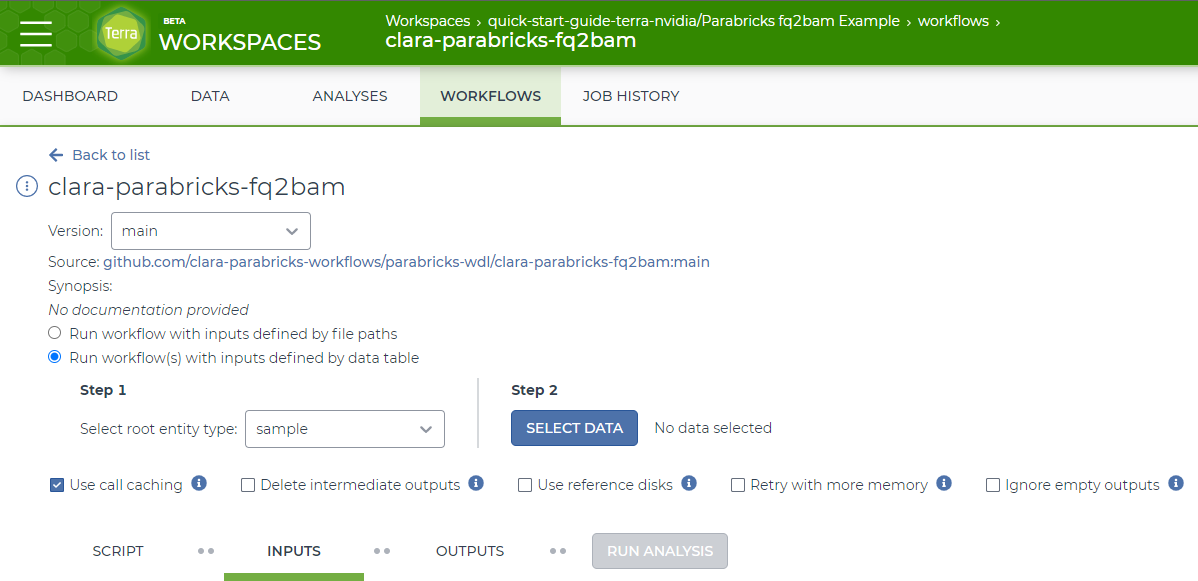
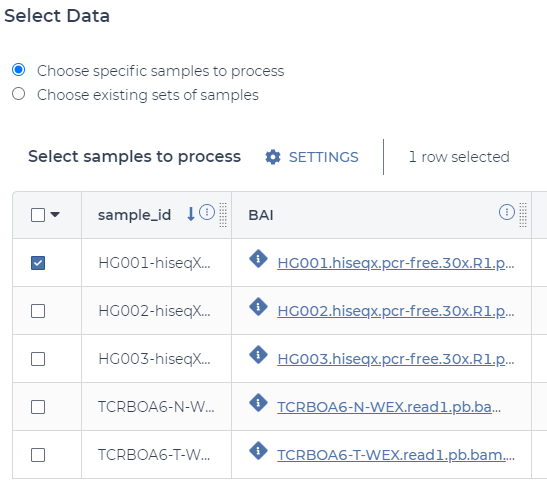
一些示例数据已为我们加载,因此我们可以单击“选择数据”以选择一个样本来运行 fq2bam 管道。在本教程中,让我们选择第一个样本。
这将使用适当的文件类型自动填充工作流程以运行 fq2bam 分析,因此我们现在要做的就是选择“运行分析”。这将带我们到一个新页面,我们可以在其中监控作业
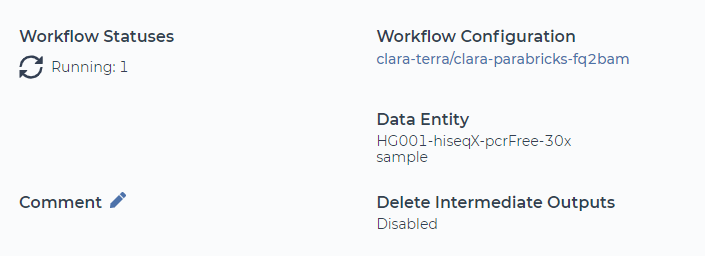
作业应该需要几分钟才能被队列接受,还需要几分钟才能完成。作业完成后,我们可以检查是否生成了输出文件。让我们点击“执行目录”。
这将带我们进入 Google Cloud 控制台,我们可以在其中查看生成的输出文件。
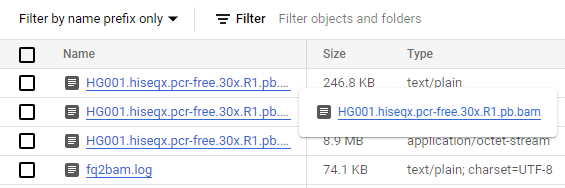
恭喜!我们现在已经使用 Terra 运行了我们的第一个 Parabricks 作业。