germline (GATK Germline Pipeline)
GATK,即 Genome Analysis Toolkit(基因组分析工具包),是由麻省理工学院和哈佛大学博德研究所开发的行业标准软件包,旨在用于广泛的基因组分析,包括变异发现、基因分型等。GATK 是生物信息学中最流行的工具之一,用于分析下一代测序数据集,并且是从种系样本中的测序数据中调用单核苷酸变异 (SNV) 和插入/缺失 (InDel) 的行业标准。
有关所有可用选项的详细列表,请参阅germline 参考部分。
GATK 提供对测序数据的强大而准确的分析,并经常更新以包含变异发现的最新最佳实践。凭借高可靠性和用于多种用例的能力,GATK 是任何使用下一代测序数据的研究人员的金标准工具。
用于变异调用的 GATK germline 工作流程可以部署在 NVIDIA 的 Parabricks 软件套件中,该套件专为加速基因组学中的二级分析而设计,将行业标准工具和工作流程从 CPU 转移到 GPU,并以高达 60 倍的速度提供相同的结果。在 NVIDIA DGX 系统上,可以在 25 分钟内分析 30 倍全基因组,而在 CPU 实例(m5.24xlarge,96 x vCPU)上则需要 30 多个小时,外显子组只需 4 分钟即可分析完成。这意味着 Parabricks 在一台 NVIDIA DGX A100 上运行,每年可以分析多达 25,000 个全基因组。NVIDIA 团队与博德研究所的 GATK 团队合作评估了 germline 工作流程的准确性。通过这一严谨的过程,他们验证了 Parabricks 工作流程产生的结果在功能上等同于 CPU 原生的 GATK 版本。
作为一个具体的例子,在使用 Parabricks 套件中的 fq2bam 和 germline caller 工作流程对公开可用的 Genome in a Bottle (GIAB) 样本进行基准测试时,产生的变异调用结果在精确度和召回率方面都 >0.9999 等同于 GATK 的全基因组 Germline 单样本变异调用工作流程中 BWA、MarkDuplicates、BQSR 和 HaplotypeCaller 命令产生的结果。
给定一对或多对 FASTQ 文件,您可以运行 germline 变异工具来生成 BAM、变异、重复指标和 recal 文件。
下面显示的 germline 流程类似于 GATK4 最佳实践流程。输入是 BWA 索引的参考文件、双端 FASTQ 文件和用于 BQSR 计算的 knownSites。此流程的输出如下:
比对、坐标排序、标记重复的 BAM 文件
BQSR 报告
vcf/g.vcf/g.vcf.gz格式的变异
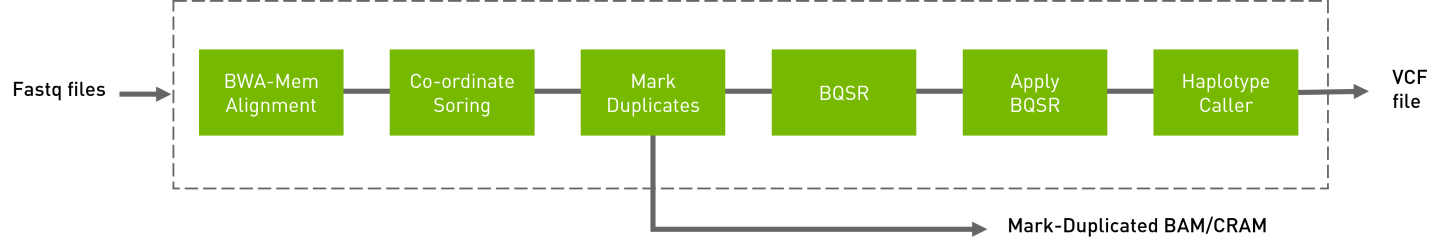
运行 germline 流程
# This command assumes all the inputs are in INPUT_DIR and all the outputs go to OUTPUT_DIR.
docker run --rm --gpus all --volume INPUT_DIR:/workdir --volume OUTPUT_DIR:/outputdir \
--workdir /workdir \
nvcr.io/nvidia/clara/clara-parabricks:4.4.0-1 \
pbrun germline \
--ref /workdir/${REFERENCE_FILE} \
--in-fq /workdir/${INPUT_FASTQ_1} /workdir/${INPUT_FASTQ_2} \
--knownSites /workdir/${KNOWN_SITES_FILE} \
--out-bam /outputdir/${OUTPUT_BAM} \
--out-variants /outputdir/${OUTPUT_VCF} \
--out-recal-file /outputdir/${OUT_RECAL_FILE}
Parabricks 支持多个原始 HaplotypeCaller 选项。要指定包含或排除多个 haplotype caller 注释,请使用 --haplotypecaller-options 选项
$ # This command assumes all the inputs are in INPUT_DIR and all the outputs go to OUTPUT_DIR.
docker run --rm --gpus all --volume INPUT_DIR:/workdir --volume OUTPUT_DIR:/outputdir \
--workdir /workdir \
nvcr.io/nvidia/clara/clara-parabricks:4.4.0-1 \
pbrun haplotypecaller \
...
--haplotypecaller-options '-min-pruning 4 -A AS_BaseQualityRankSumTest -A TandemRepeat'
...
可以使用 -AX 选项以相同方式排除注释。-A/-AX 标志及其值之间应有一个空格。
以下是支持的选项及其允许的值
- -A
AS_BaseQualityRankSumTest
AS_FisherStrand
AS_InbreedingCoeff
AS_MappingQualityRankSumTest
AS_QualByDepth
AS_RMSMappingQuality
AS_ReadPosRankSumTest
AS_StrandOddsRatio
AssemblyComplexity
BaseQualityRankSumTest
ChromosomeCounts
ClippingRankSumTest
Coverage
DepthPerAlleleBySample
DepthPerSampleHC
ExcessHet
FisherStrand
InbreedingCoeff
MappingQualityRankSumTest
QualByDepth
RMSMappingQuality
ReadPosRankSumTest
ReferenceBases
StrandBiasBySample
StrandOddsRatio
TandemRepeat
- -AX
(与 -A 选项相同)
- --output-mode
EMIT_VARIANTS_ONLY
EMIT_ALL_CONFIDENT_SITES
EMIT_ALL_ACTIVE_SITES
- -max-reads-per-alignment-start
一个正整数
- -min-dangling-branch-length
一个正整数
- -min-pruning
一个正整数
- -pcr-indel-model
NONE
HOSTILE
AGGRESSIVE
CONSERVATIVE
- -standard-min-confidence-threshold-for-calling
一个正整数
以下命令是上述 Parabricks 命令的 bwa-0.7.12 和 GATK4 对等命令。这些命令的输出将与上述命令的输出相同。有关比较结果,请参阅输出比较页面。
# Run bwa-mem and pipe output to create sorted BAM
$ bwa mem \
-t 32 \
-K 10000000 \
-R '@RG\tID:sample_rg1\tLB:lib1\tPL:bar\tSM:sample\tPU:sample_rg1' \
<INPUT_DIR>/${REFERENCE_FILE} <INPUT_DIR>/${INPUT_FASTQ_1} <INPUT_DIR>/${INPUT_FASTQ_2} | \
gatk SortSam \
--java-options -Xmx30g \
--MAX_RECORDS_IN_RAM 5000000 \
-I /dev/stdin \
-O cpu.bam \
--SORT_ORDER coordinate
# Mark Duplicates
$ gatk MarkDuplicates \
--java-options -Xmx30g \
-I cpu.bam \
-O mark_dups_cpu.bam \
-M metrics.txt
# Generate BQSR Report
$ gatk BaseRecalibrator \
--java-options -Xmx30g \
--input mark_dups_cpu.bam \
--output <OUTPUT_DIR>/${OUT_RECAL_FILE} \
--known-sites <INPUT_DIR>/${KNOWN_SITES_FILE} \
--reference <INPUT_DIR>/${REFERENCE_FILE}
# Run ApplyBQSR Step
$ gatk ApplyBQSR \
--java-options -Xmx30g \
-R <INPUT_DIR>/${REFERENCE_FILE} \
-I mark_dups_cpu.bam \
--bqsr-recal-file <OUTPUT_DIR>/${OUT_RECAL_FILE} \
-O cpu_nodups_BQSR.bam
#Run Haplotype Caller
$ gatk HaplotypeCaller \
--java-options -Xmx30g \
--input cpu_nodups_BQSR.bam \
--output <OUTPUT_DIR>/${OUTPUT_VCF} \
--reference <INPUT_DIR>/${REFERENCE_FILE} \
--native-pair-hmm-threads 16
运行 Germline 流程以将 FASTQ 转换为 VCF。
输入/输出文件选项
- --ref REF
-
参考文件的路径。(默认值:None)
此选项为必选项。
- --in-fq [IN_FQ ...]
-
双端 FASTQ 文件的路径,后跟带引号的可选读取组(示例:“@RG\tID:foo\tLB:lib1\tPL:bar\tSM:sample\tPU:foo”)。文件必须为 fastq 或 fastq.gz 格式。所有输入集都应具有读取组;否则,都不应具有读取组,并且管道将自动添加读取组。此选项可以重复多次。示例 1:--in-fq sampleX_1_1.fastq.gz sampleX_1_2.fastq.gz --in-fq sampleX_2_1.fastq.gz sampleX_2_2.fastq.gz。示例 2:--in-fq sampleX_1_1.fastq.gz sampleX_1_2.fastq.gz "@RG\tID:foo\tLB:lib1\tPL:bar\tSM:sample\tPU:unit1" --in-fq sampleX_2_1.fastq.gz sampleX_2_2.fastq.gz "@RG\tID:foo2\tLB:lib1\tPL:bar\tSM:sample\tPU:unit2"。对于同一示例,读取组应具有相同的样本名称 (SM) 和不同的 ID 和 PU。(默认值:None)
- --in-se-fq [IN_SE_FQ ...]
-
单端 FASTQ 文件的路径,后跟带引号的可选读取组(示例:“@RG\tID:foo\tLB:lib1\tPL:bar\tSM:sample\tPU:foo”)。文件必须为 fastq 或 fastq.gz 格式。所有输入集都应具有读取组,或者都不应具有读取组,并且管道将自动添加读取组。此选项可以重复多次。示例 1:--in-se-fq sampleX_1.fastq.gz --in-se-fq sampleX_2.fastq.gz 。示例 2:--in-se-fq sampleX_1.fastq.gz "@RG\tID:foo\tLB:lib1\tPL:bar\tSM:sample\tPU:unit1" --in-se-fq sampleX_2.fastq.gz "@RG\tID:foo2\tLB:lib1\tPL:bar\tSM:sample\tPU:unit2" 。对于同一示例,读取组应具有相同的样本名称 (SM) 和不同的 ID 和 PU。(默认值:None)
- --knownSites KNOWNSITES
-
已知 indels 文件的路径。文件必须为 vcf.gz 格式。此选项可以多次使用。(默认值:None)
- --interval-file INTERVAL_FILE
-
以下格式之一的 interval 文件的路径:Picard 样式(.interval_list 或 .picard)、GATK 样式(.list 或 .intervals)或 BED 文件 (.bed)。此选项可以多次使用。(默认值:None)
- --out-recal-file OUT_RECAL_FILE
-
基础质量分数重新校准后报告文件的路径。(默认值:None)
- --out-bam OUT_BAM
-
标记重复项后 BAM 文件的路径。(默认值:None)
此选项为必选项。
- --htvc-bam-output HTVC_BAM_OUTPUT
-
应在 HaplotypeCaller 中写入程序集单倍型的文件。如果使用 --run-partition 传递,则将输出多个 BAM 文件。(默认值:None)
- --out-variants OUT_VARIANTS
-
变异调用后 vcf/gvcf/gvcf.gz 文件的路径。(默认值:None)
此选项为必选项。
- --out-duplicate-metrics OUT_DUPLICATE_METRICS
-
标记重复项后重复指标文件的路径。(默认值:None)
- --htvc-alleles HTVC_ALLELES
-
vcf.gz force-call 文件的路径。要强制调用的等位基因集,而无需考虑证据。(默认值:None)
工具选项
- --max-read-length MAX_READ_LENGTH
-
用于 bwa 和筛选 FASTQ 输入的最大读取长度/大小(即序列长度)(默认值:480)
- --min-read-length MIN_READ_LENGTH
-
用于 bwa 和筛选 FASTQ 输入的最小读取长度/大小(即序列长度)(默认值:1)
- -L INTERVAL, --interval INTERVAL
-
从中调用输入读取的 bqsr 的 Interval。所有 interval 都将具有 100 的填充以获取读取记录,并且重叠的 interval 将被组合。Interval 文件应使用 --interval-file 选项传递。此选项可以多次使用(例如“-L chr1 -L chr2:10000 -L chr3:20000+ -L chr4:10000-20000”)。(默认值:None)
- --bwa-options BWA_OPTIONS
-
将支持的 bwa mem 选项作为字符串传递。当前原始 bwa mem 支持的选项为:-M、-Y、-C、-T、-B、-U、-L 和 -K(例如 --bwa-options="-M -Y")(默认值:None)
- --no-warnings
-
禁止显示有关系统线程和内存使用情况的警告消息。(默认值:None)
- --filter-flag FILTER_FLAG
-
如果条目的标志满足此条件,则不要在输出中生成 SAM 条目。条件:(flag & filter != 0)(默认值:0)
- --skip-multiple-hits
-
筛选 SA 长度不为 0 的 SAM 条目(默认值:None)
- --align-only
-
在 bwa-mem 之后生成输出 BAM。输出将不会进行坐标排序,也不会标记重复项(默认值:None)
- --no-markdups
-
不执行标记重复项步骤。在排序后返回 BAM。(默认值:None)
- --fix-mate
-
向输出文件添加配对雪茄 (MC) 和配对质量 (MQ) 标签。(默认值:None)
- --markdups-assume-sortorder-queryname
-
假定读取按 queryname 排序以进行标记重复项。这也将标记辅助、补充和未比对的读取为重复项。此标志不会影响变异调用,但会增加处理时间。(默认值:None)
- --markdups-picard-version-2182
-
假定标记重复项与 Picard 版本 2.18.2 类似。(默认值:None)
- --monitor-usage
-
在执行期间监视近似的 CPU 利用率和主机内存使用情况。(默认值:None)
- --optical-duplicate-pixel-distance OPTICAL_DUPLICATE_PIXEL_DISTANCE
-
两个重复簇之间的最大偏移量,以便将其视为光学重复项。如果未传递 --out-duplicate-metrics,则忽略此选项。(默认值:None)
- --read-group-sm READ_GROUP_SM
-
此运行中读取组的 SM 标签。(默认值:None)
- --read-group-lb READ_GROUP_LB
-
此运行中读取组的 LB 标签。(默认值:None)
- --read-group-pl READ_GROUP_PL
-
此运行中读取组的 PL 标签。(默认值:None)
- --read-group-id-prefix READ_GROUP_ID_PREFIX
-
此运行中读取组的 ID 和 PU 标签的前缀。此前缀将用于此运行中的所有 FASTQ 文件对。ID 和 PU 标签将包含此前缀和一个标识符,该标识符对于一对 FASTQ 文件是唯一的。(默认值:None)
- -ip INTERVAL_PADDING, --interval-padding INTERVAL_PADDING
-
要添加到您包含的每个 interval 的填充量(以碱基对为单位)。(默认值:None)
- --standalone-bqsr
-
运行独立的 BQSR。(默认值:None)
- --haplotypecaller-options HAPLOTYPECALLER_OPTIONS
-
将支持的 haplotype caller 选项作为字符串传递。以下是当前支持的原始 haplotypecaller 选项:-A <AS_BaseQualityRankSumTest、AS_FisherStrand、AS_InbreedingCoeff、AS_MappingQualityRankSumTest、AS_QualByDepth、AS_RMSMappingQuality、AS_ReadPosRankSumTest、AS_StrandOddsRatio、BaseQualityRankSumTest、ChromosomeCounts、ClippingRankSumTest、Coverage、DepthPerAlleleBySample、DepthPerSampleHC、ExcessHet、FisherStrand、InbreedingCoeff、MappingQualityRankSumTest、QualByDepth、RMSMappingQuality、ReadPosRankSumTest、ReferenceBases、StrandBiasBySample、StrandOddsRatio、TandemRepeat、AssemblyComplexity>、-AX <与 -A 相同的选项>、--output-mode <EMIT_VARIANTS_ONLY、EMIT_ALL_CONFIDENT_SITES、EMIT_ALL_ACTIVE_SITES>、-max-reads-per-alignment-start <int>、-min-dangling-branch-length <int>、-min-pruning <int>、-pcr-indel-model <NONE、HOSTILE、AGGRESSIVE、CONSERVATIVE>、-standard-min-confidence-threshold-for-calling <int>(例如 --haplotypecaller-options="-min-pruning 4 -standard-min-confidence-threshold-for-calling 30")。(默认值:None)
- --static-quantized-quals STATIC_QUANTIZED_QUALS
-
使用静态量化质量分数到给定数量的级别。为多个 bin 重复此选项多次。(默认值:None)
- --gvcf
-
以 .gvcf 格式生成变异调用。(默认值:None)
- --disable-read-filter DISABLE_READ_FILTER
-
禁用 BAM 条目的读取过滤器。当前,可以禁用的受支持读取过滤器为 MappingQualityAvailableReadFilter、MappingQualityReadFilter、NotSecondaryAlignmentReadFilter 和 WellformedReadFilter。(默认值:None)
- --max-alternate-alleles MAX_ALTERNATE_ALLELES
-
要进行基因分型的最大备择等位基因数。(默认值:None)
- -G ANNOTATION_GROUP, --annotation-group ANNOTATION_GROUP
-
要添加到输出变异调用的注释组。当前支持的注释组为 StandardAnnotation、StandardHCAnnotation 和 AS_StandardAnnotation。(默认值:None)
- -GQB GVCF_GQ_BANDS, --gvcf-gq-bands GVCF_GQ_BANDS
-
参考置信度 GQ 波段的独占上限。必须在 [1, 100] 范围内,并按递增顺序指定。(默认值:None)
- --rna
-
运行针对 RNA 数据优化的 haplotypecaller。(默认值:None)
- --dont-use-soft-clipped-bases
-
请勿使用 soft clipped 碱基进行变异调用。(默认值:None)
- --minimum-mapping-quality MINIMUM_MAPPING_QUALITY
-
要保留的最小比对质量(含)。(默认值:None)
- --mapping-quality-threshold-for-genotyping MAPPING_QUALITY_THRESHOLD_FOR_GENOTYPING
-
在活动区域检测和程序集步骤之后但在基因分型之前,控制用于从基因分型器中排除由于比对质量不佳的读取的阈值。(默认值:None)
- --enable-dynamic-read-disqualification-for-genotyping
-
将启用不太严格的读取排除低碱基质量读取。(默认值:None)
- --min-base-quality-score MIN_BASE_QUALITY_SCORE
-
考虑用于调用的碱基所需的最小碱基质量。(默认值:None)
- --adaptive-pruning
-
在修剪 De Bruijn 图时使用自适应图修剪算法。(默认值:None)
- --force-call-filtered-alleles
-
强制调用 --alleles 指定的资源中包含的已筛选等位基因。(默认值:None)
- --filter-reads-too-long
-
忽略大小 > 500bp 的所有输入 BAM 读取(默认值:None)
- --no-alt-contigs
-
去除备择 contig 的输出记录。(默认值:None)
- --ploidy PLOIDY
-
BAM 文件假定的倍性。当前仅支持单倍体(倍性 1)和二倍体(倍性 2)。(默认值:2)
- --sample-sex SAMPLE_SEX
-
样本输入的性别。此选项将覆盖从任何 X/Y 读取比率范围确定的性别。必须为男性或女性。(默认值:None)
- --range-male RANGE_MALE
-
X/Y 读取比率的内含男性范围。如果实际比率落在指定范围内,则将性别声明为男性。语法为“<min>-<max>”(例如“--range-male 1-10”)。(默认值:None)
- --range-female RANGE_FEMALE
-
X/Y 读取比率的内含女性范围。如果实际比率落在指定范围内,则将性别声明为女性。语法为“<min>-<max>”(例如“--range-female 150-250”)。(默认值:None)
- --use-GRCh37-regions
-
对 GRCh37 参考类型使用假常染色体区域。此标志应与 GRCh37 和 UCSC hg19 参考一起使用。默认情况下,使用 GRCh38 区域。
(默认值:None)
性能选项
- --bwa-nstreams BWA_NSTREAMS
-
每个 GPU 要使用的流数;注意:更多流会增加设备内存使用量(默认值:4)
- --bwa-cpu-thread-pool BWA_CPU_THREAD_POOL
-
每个 GPU 要专用于 CPU 线程池的线程数(默认值:16)
- --num-cpu-threads-per-stage NUM_CPU_THREADS_PER_STAGE
-
(与上述相同)每个 GPU 要专用于 CPU 线程池的线程数(默认值:None)
- --gpuwrite
-
使用一个 GPU 加速写入最终 BAM/CRAM。(默认值:None)
- --gpuwrite-deflate-algo GPUWRITE_DEFLATE_ALGO
-
选择要与 --gpuwrite 一起使用的 nvCOMP DEFLATE 算法。请注意,这些选项与 CPU DEFLATE 选项不对应。有效选项为 1、2 和 4。选项 1 最快,而选项 2 和 4 的吞吐量逐渐降低,但压缩率更高。当用户未提供输入时(即 None),默认值为 1(默认值:None)
- --gpusort
-
使用 GPU 加速排序和标记。(默认值:None)
- --use-gds
-
使用 GPUDirect Storage (GDS) 来启用直接数据路径,用于 GPU 内存和存储之间的直接内存访问 (DMA) 传输。必须与 --gpuwrite 同时使用。有关如何设置和使用 GPUDirect Storage 的信息,请参阅 Parabricks 文档 > 最佳性能。(默认值:None)
- --memory-limit MEMORY_LIMIT
-
排序和后排序期间以 GB 为单位的系统内存限制。默认情况下,限制为系统总内存的一半。(默认值:62)
- --low-memory
-
使用低内存模式;将降低每个 GPU 的流数(默认值:None)
- --htvc-low-memory
-
在 htvc 中使用低内存模式。(默认值:None)
- --num-htvc-threads NUM_HTVC_THREADS
-
CPU 线程数。(默认值:5)
- --run-partition
-
将整个基因组划分为多个分区,并同时运行多个进程,每个进程在一个分区上运行。(默认值:None)
- --gpu-num-per-partition GPU_NUM_PER_PARTITION
-
每个分区要使用的 GPU 数量。(默认值:None)
- --read-from-tmp-dir
-
运行变异调用程序,从 Aligner 和 sort 生成的 bin 文件中读取。并行运行 postsort。此选项将增加设备内存使用量。(默认值:None)
通用选项
- --logfile LOGFILE
-
日志文件的路径。如果未指定,消息将仅写入标准错误输出。(默认值:None)
- --tmp-dir TMP_DIR
-
将存储临时文件的目录的完整路径。
- --with-petagene-dir WITH_PETAGENE_DIR
-
PetaGene 安装目录的完整路径。默认情况下,这应已安装在 /opt/petagene 中。使用此选项还需要通过设置 LD_PRELOAD 环境变量来预加载 PetaLink 库。可以选择设置用于数据和凭据的 PETASUITE_REFPATH 和 PGCLOUD_CREDPATH 环境变量(默认值:None)
- --keep-tmp
-
完成时不删除存储临时文件的目录。
- --no-seccomp-override
-
不覆盖 docker 的 seccomp 选项(默认值:None)。
- --version
-
查看兼容的软件版本。
GPU 选项
- --num-gpus NUM_GPUS
-
运行要使用的 GPU 数量。将使用 GPU 0..(NUM_GPUS-1)。
--in-fq 选项采用两个 FASTQ 文件的名称,可以选择后跟带引号的读取组。FASTQ 文件名不得以连字符开头。
在提供给 --haplotypecaller-options 的值中,--output-mode 需要两个前导连字符,而所有其他值都采用单个连字符。