在 AWS 上使用 Parabricks 对 Complete Genomics 的全基因组和全外显子组数据进行基准测试
这是一个使用 Complete Genomics 测序仪的数据对 Parabricks 生殖系工作流程进行基准测试的快速入门指南。Parabricks 是一个用于基因组学二级分析的 GPU 加速工具包。在本指南中,我们将展示 Parabricks 如何在使用来自 Complete Genomics 的 DNBSEQ-T7 和 DNBSEQ-G400 测序仪的数据在云端以快速且经济高效的方式运行。
基因组文件(如 FASTQ 和 BAM 文件)很容易达到每个数百 GB 的大小。当运行涉及数十万个此类文件的研究时,很容易达到 TB 级的数据量,并且处理所有这些数据的成本变得非常高昂。当在云端运行时,这一点尤其明显,因为用户按小时付费,因此每一分钟的计算都很重要。我们处理数据的速度越快,成本就越低。
本示例随附的代码可以在 GitHub 上找到。
软件
这些基准测试是使用 Parabricks 4.0.1-1 版本执行的,该版本作为 Docker 容器在 NVIDIA GPU 云 (NGC) 上公开提供,可通过运行以下命令获得
docker pull nvcr.io/nvidia/clara/clara-parabricks:4.0.1-1
其他软件先决条件包括
软件 |
版本 |
用途 |
|---|---|---|
| bwa | 0.7.18 | 索引参考基因组 |
| seqtk | 1.4 | 下采样 FASTQ |
为了最大限度地提高 Parabricks 的性能,最好所有文件读取和写入都在机器上的快速 SSD 上进行。要查找 SSD 的路径,请运行
lsblk
导出挂载点作为环境变量 NVME_DRIVE,以便此存储库中的脚本知道从哪里下载和运行数据
export NVME_DRIVE=/opt/dlami/nvme
硬件
对于 Parabricks,我们推荐两种 GPU 类别:高性能 GPU(A100、H100、L40S)和低成本 GPU(A10、L4)。这些基准测试已使用 AWS 上的 L40S 实例和 L4 实例进行了验证,但任何类似配置的机器都可以工作。请务必查看 Parabricks 文档以了解最低要求。以下是我们验证中使用的确切配置
配置 |
L4 |
L40S |
|---|---|---|
| 实例类型 | g6.24xlarge | g6e.24xlarge |
| 操作系统 | Ubuntu | Ubuntu |
| AMI | 深度学习 Nvidia 驱动程序 | 深度学习 Nvidia 驱动程序 |
| GPU 数量 | 4 | 4 |
| vCPU 数量 | 96 | 96 |
| CPU 内存 | 384 GB | 768 GB |
| 按需每小时成本* | $6.68 | $15.07 |
对于这些基准测试,我们将使用来自 DNBSEQ-T7 和 DNBSEQ-G400 Complete Genomics 测序仪的 NA12878 全基因组 (WGS) 数据。所有数据,包括 FASTQ、参考基因组和其他辅助文件,都公开托管,可以使用以下命令下载
download.sh
预处理
公开存在的数据几乎可以直接用于我们的基准测试。为了进行同类比较,我们希望两个 WGS 样本都具有相同的覆盖率。T7 WGS 数据的覆盖率为 46 倍,G400 WGS 数据的覆盖率为 30 倍。为了解决这个问题,我们将 T7 WGS 数据下采样 65%。为了实现这一点,我们可以运行
downsample.sh 0.65
这将重命名 E100030471QC960_L01_48_1.fq.gz 为 E100030471QC960_L01_48_1.30x.fq.gz,然后数据就可以通过基准测试运行了。
下表总结了每个样本的最终覆盖率、读取次数和文件大小
T7 |
G400 |
|
|---|---|---|
| 30 倍 | 30 倍 | |
| 6.58 亿 | 9.55 亿 | |
| 72 GB | 69 GB |
查看 benchmarks 文件夹将显示可用的基准测试脚本
benchmarks/
├── L4
│ ├── deepvariant.sh
│ └── germline.sh
└── L40S
├── deepvariant.sh
└── germline.sh
对于每组硬件,都有一个 germline(生殖系)和一个 deepvariant 脚本。这种分离是由于每种配置使用不同的优化标志。要了解有关这些优化的更多信息,请查看 Parabricks 文档 。germline 脚本运行 Parabricks 生殖系流程,该流程对 FASTQ 文件进行比对并运行 HaplotypeCaller。deepvariant 脚本运行 Parabricks deepvariant 变异调用器。
benchmark.sh 脚本接受一个参数,用于指定硬件类型,该参数与 benchmarks 文件夹中的文件夹名称匹配。例如,要运行 L4 基准测试,我们可以运行
./benchmarks L4
类似地,要运行 L40S 基准测试,我们可以运行
./benchmarks L40S
Parabricks 软件会输出管道中每个步骤的运行时间,这些数字已记录在下表中。运行基准测试后,运行时间将捕获在位于 ${NVME_DIR}/data/logs 的日志文件中。下图是每个 GPU 类型上每个样本的运行时间图。
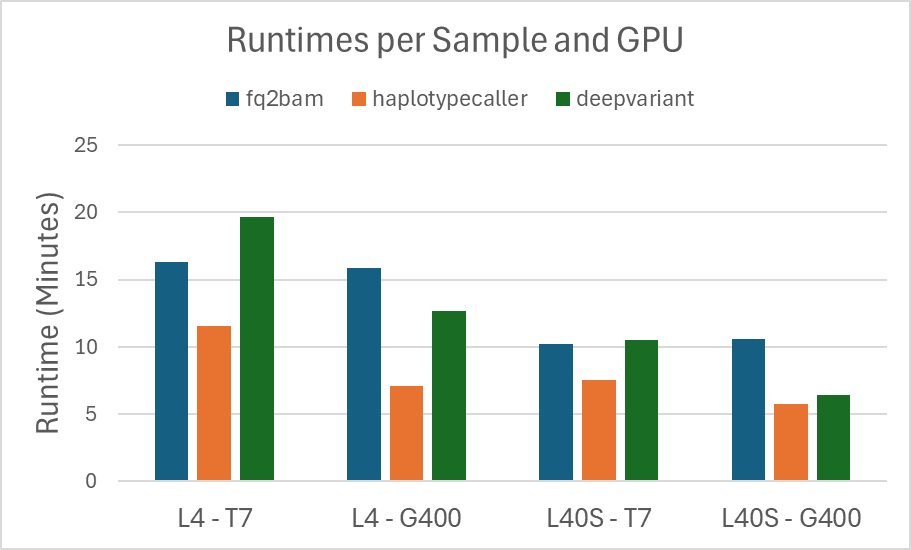
图 1. fq2bam、haplotypecaller 和 deepvariant 在每个 GPU 类型上每个样本的运行时间。
正如我们所预期的那样,对于每个样本,L40S 实例的运行时间都比 L4 实例的运行时间快。这种差异也反映在成本上,如下面的图表所示
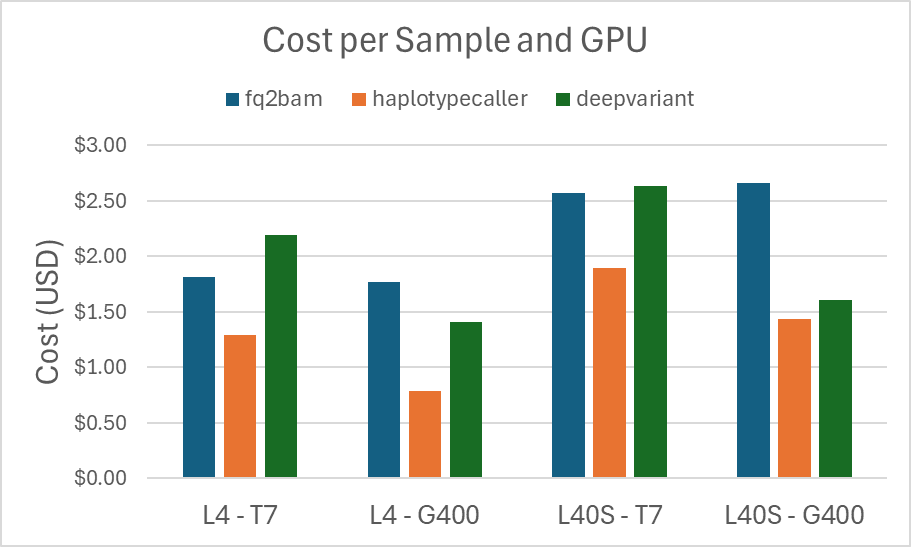
图 2. fq2bam、haplotypecaller 和 deepvariant 在每个 GPU 类型上每个样本的成本。
用于这些图表的精确数字汇总在下表中。
L4 |
L40S |
|||||||
|---|---|---|---|---|---|---|---|---|
T7 |
G400 |
T7 |
G400 |
|||||
运行时间 |
成本 |
运行时间 |
成本 |
运行时间 |
成本 |
运行时间 |
成本 |
|
| fq2bam | 17 | $1.85 | 16 | $1.77 | 10 | $2.39 | 11 | $2.66 |
| haplotypecaller | 7 | $0.82 | 7 | $0.68 | 6 | $1.44 | 6 | $1.44 |
| deepvariant | 13 | $1.44 | 13 | $1.41 | 7 | $1.70 | 6 | $1.60 |
除了运行时间数字外,重要的是检查变异的质量是否与真值集非常匹配。
NA12878 基本事实 VCF 可以在 NIH FTP 上找到。由于此 VCF 是使用 GRCh37 参考基因组运行的,但我们的样本是使用 UCSC hg19 参考基因组运行的,因此我们首先需要进行 liftover,然后才能运行一致性分析。
作为可选步骤,可以使用 liftover.sh 和 concordance.sh 脚本分别完成此操作。
下表是我们使用此工作流程实现的结果
DeepVariant 一致性 |
|||||||
|---|---|---|---|---|---|---|---|
| 类型 | 过滤器 | TRUTH.TP | TRUTH.FN | QUERY.FP | METRIC.召回率 | METRIC.精确率 | METRIC_F1_Score |
| INDEL | 全部 | 460832 | 6052 | 1389 | 0.987037 | 0.997107 | 0.992047 |
| INDEL | 通过 | 460832 | 6052 | 1389 | 0.987037 | 0.997107 | 0.992047 |
| SNP | 全部 | 3213043 | 38814 | 6106 | 0.988064 | 0.998104 | 0.993059 |
| SNP | 通过 | 3213043 | 38814 | 6106 | 0.988064 | 0.998104 | 0.993059 |
DeepVariant 一致性 |
|||||||
|---|---|---|---|---|---|---|---|
| 类型 | 过滤器 | TRUTH.TP | TRUTH.FN | QUERY.FP | METRIC.召回率 | METRIC.精确率 | METRIC_F1_Score |
| INDEL | 全部 | 460832 | 6052 | 1389 | 0.987037 | 0.997107 | 0.992047 |
| INDEL | 通过 | 460832 | 6052 | 1389 | 0.987037 | 0.997107 | 0.992047 |
| SNP | 全部 | 3213043 | 38814 | 6106 | 0.988064 | 0.998104 | 0.993059 |
| SNP | 通过 | 3213043 | 38814 | 6106 | 0.988064 | 0.998104 | 0.993059 |
DeepVariant 一致性 |
|||||||
|---|---|---|---|---|---|---|---|
| 类型 | 过滤器 | TRUTH.TP | TRUTH.FN | QUERY.FP | METRIC.召回率 | METRIC.精确率 | METRIC_F1_Score |
| INDEL | 全部 | 459953 | 6931 | 2320 | 0.985155 | 0.995165 | 0.990135 |
| INDEL | 通过 | 459953 | 6931 | 2320 | 0.985155 | 0.995165 | 0.990135 |
| SNP | 全部 | 3208100 | 43757 | 7435 | 0.986544 | 0.997689 | 0.992085 |
| SNP | 通过 | 3208100 | 43757 | 7435 | 0.986544 | 0.997689 | 0.992085 |
DeepVariant 一致性 |
|||||||
|---|---|---|---|---|---|---|---|
| 类型 | 过滤器 | TRUTH.TP | TRUTH.FN | QUERY.FP | METRIC.召回率 | METRIC.精确率 | METRIC_F1_Score |
| INDEL | 全部 | 459224 | 7660 | 5812 | 0.983593 | 0.987961 | 0.985772 |
| INDEL | 通过 | 459224 | 7660 | 5812 | 0.983593 | 0.987961 | 0.985772 |
| SNP | 全部 | 3199856 | 52001 | 47560 | 0.984009 | 0.985358 | 0.984683 |
| SNP | 通过 | 3199856 | 52001 | 47560 | 0.984009 | 0.985358 | 0.984683 |
正如我们对 Parabricks 的期望一样,我们看到精确率和 F1 分数都超过 99%,这证实了变异调用器相对于基本事实是准确的。
在本指南中,我们展示了如何从 Complete Genomics 下载 WGS 数据,在 AWS 上运行比对 (fq2bam) 和变异调用 (haplotypecaller 和 deepvariant),显示每个样本的运行时间和总成本,最后演示了针对真值集的一致性结果。总而言之,这表明 Parabricks 支持来自 Complete Genomics 测序仪的数据,因为它在这些工具上运行快速且准确。