全基因组体细胞小变异检测
本操作指南探讨如何使用 Mutect2 在 SEQC2 联盟样本中进行变异检测。
Samtools 和 BWA
NVIDIA Parabricks
GATK4 v4.2.0.0
bedtools
R (vcfR, UpSetR)
至少 512GB 的磁盘空间(最好是“平衡”或 SSD 存储)
至少两个 NVIDIA Tesla T4 GPU;以下命令和时间估计使用四个 NVIDIA Tesla T4 GPU。
至少 24 个 CPU 核心;以下命令使用 48 个 vCPU 核心。
每个 GPU 至少 48GB 的 RAM
功能正常的 Parabricks 安装(4.0 或更新版本)
以下示例使用 AWS VM g4dn.12xlarge,配备 48 个 vCPU 核心、192GB RAM 和 4 个 NVIDIA Tesla T4 GPU,运行 Ubuntu 18.04.5 LTS。
本操作指南完整介绍了全基因组测序 (WGS) 体细胞变异分析流程,用于在真实的 30X 短读人数据上检测 SNP、MNP 和小插入缺失。此类分析常用于癌症基因组学研究。对于 WGS 体细胞变异分析,您将使用“SEQC2 联盟体细胞突变工作组”生成的示例数据。该数据集包含来自 HCC1395(一种三阴性乳腺癌细胞系)和 HCC1395 BL(匹配的正常细胞系)的多平台测序数据。SEQC2 数据集包含在六个不同测序中心进行的整个基因组 (WGS) 和外显子测序,以及多个重复,以最大限度地减少来自测序技术/分析的潜在偏差。此外,该数据集通过九个生物信息学流程进行处理,以评估准确性和可重复性。SEQC2 联盟报告了由于样本和文库处理产生的伪影,并评估了生物信息学工具在伪影检测和去除方面的能力和局限性。以下提供了一系列详细文章
SEQC2 数据集具有良好的特征,联盟提供的“真实变异调用集”允许您验证 WGS 体细胞变异分析流程的结果。
在变异检测之后,您需要使用一个或多个数据库注释 VCF,以确定哪些变异是常见的或与疾病相关的(例如在 1000 基因组中频繁观察到的变异),过滤掉那些常见的变异,然后使用 Parabricks 工具进行质量控制,以评估变异检测器的结果。本操作指南不涵盖注释或过滤。
本操作指南使用的示例数据集位于 NCBI SRA 上,可以使用 wget 下载。下载 SRA 文件后,安装 SRA Toolkit 以使用以下说明将 SRA 文件转换为配对末端 FASTQ 文件。请注意,SRR7890827 是正常样本,SRR7890824 是来自 HCC1395 细胞系的肿瘤样本。每个样本都在 Illumina HiSeq X 上进行了配对末端 WGS 测序。
## Download publicly available SRA files using wget. Both files are
# about 65 GB in size.
# Normal sample
$ wget https://sra-pub-run-odp.s3.amazonaws.com/sra/SRR7890827/SRR7890827 --output-document=SRR7890827.sra
# Tumor sample
$ wget https://sra-pub-run-odp.s3.amazonaws.com/sra/SRR7890824/SRR7890824 --output-document=SRR7890824.sra
## Convert SRA to FASTQ files
$ fastq-dump --split-files ./SRR7890827.sra --gzip
$ fastq-dump --split-files ./SRR7890824.sra --gzip
SRA 文件转换为 FASTQ 格式后,不再需要,可以删除。
如果您有自己的数据,您很可能可以直接将其替换到本操作指南中的命令中,以获得准确的、多调用器的变异调用。
接下来,下载并索引参考基因组,用于比对和变异检测。您将使用 GRCh38 进行比对,可以从此页面下载
从该网站下载并解压以下内容
GRCh38.d1.vd1.fa.tar.gz
如果您不想构建自己的 BWA 索引,还可以下载并解压以下内容
GRCh38.d1.vd1_BWA.tar.gz 和
GRCh38.d1.vd1_GATK_indices.tar.gz
否则,您需要构建自己的索引。构建索引的说明在下方给出。
您将需要这些额外的 VCF 文件及其索引
此外,您还需要真值集的 BED 和 VCF 文件
如果您使用的参考文件没有可用的索引,您可以使用 BWA 对其进行索引。索引编制应耗时 30 到 60 分钟。
$ tar -xvzf GRCh38.d1.vd1.fa.tar.gz
## Note this is baseline bwa.
$ bwa index GRCh38.d1.vd1.fa
pbrun fq2bam 命令运行读取比对,以及排序、重复标记和碱基质量分数重校准 (BQSR),这符合 GATK 最佳实践,但速度比社区工具快得多,因为它最多可以利用八个 NVIDIA GPU。pbrun fq2bam 命令还会生成 BQSR 报告,该报告用于提高 BAM 文件中的碱基质量,并供下游 MuTect2 用于变异检测。请注意,本操作指南添加了自定义读取组,以便更容易识别样本。如果样本稍后在下游分析中与其他样本合并,则在为匹配的肿瘤/正常或多样本分析运行比对时,添加自定义读取组至关重要。您还需要传递一组已知位点以生成 BQSR 报告
GCF_000001405.39.gz
ALL.wgs.1000G_phase3.GRCh38.ncbi_remapper.20150424.shapeit2_indels.vcf.gz
Mills_and_1000G_gold_standard.indels.b38.primary_assembly.vcf.gz
## Change the extension name to be compatible with pbrun
$ mv GCF_000001405.39.gz GCF_000001405.39.vcf.gz
## Aligning Normal sample FASTQ file
$ docker run \
--gpus all \
--workdir /workdir \
--rm \
--volume $(pwd):/workdir \
--volume $(pwd):/outputdir \
nvcr.io/nvidia/clara/clara-parabricks:4.4.0-1 \
pbrun fq2bam \
--ref /workdir/GRCh38.d1.vd1.fa \
--in-fq /workdir/SRR7890827_1.fastq.gz /workdir/SRR7890827_2.fastq.gz "@RG\tID:id_SRR7890827_rg1\tLB:lib1\tPL:bar\tSM:sm_SRR7890827\tPU:pu_SRR7890827_rg1" \
--knownSites /workdir/Mills_and_1000G_gold_standard.indels.b38.primary_assembly.vcf.gz \
--knownSites /workdir/GCF_000001405.vcf.39.gz \
--knownSites /workdir/ALL.wgs.1000G_phase3.GRCh38.ncbi_remapper.20150424.shapeit2_indels.vcf.gz \
--out-recal-file /outputdir/SRR7890827-WGS_FD_N_BQSR_REPORT.txt \
--bwa-options=-Y \
--out-bam /outputdir/SRR7890827-WGS_FD_N.bam
## Aligning Tumor sample FASTQ file
$ docker run \
--gpus all \
--workdir /workdir \
--rm \
--volume $(pwd):/workdir \
--volume $(pwd):/outputdir \
nvcr.io/nvidia/clara/clara-parabricks:4.4.0-1 \
pbrun fq2bam \
--ref /workdir/GRCh38.d1.vd1.fa \
--in-fq /workdir/SRR7890824_1.fastq.gz /workdir/SRR7890824_2.fastq.gz "@RG\tID:id_SRR7890824_rg1\tLB:lib1\tPL:bar\tSM:sm_SRR7890824\tPU:pu_SRR7890824_rg1" \
--knownSites /workdir/Mills_and_1000G_gold_standard.indels.b38.primary_assembly.vcf.gz \
--knownSites /workdir/GCF_000001405.vcf.39.gz \
--knownSites /workdir/ALL.wgs.1000G_phase3.GRCh38.ncbi_remapper.20150424.shapeit2_indels.vcf.gz \
--out-recal-file /outputdir/SRR7890824-WGS_FD_T_BQSR_REPORT.txt \
--bwa-options=-Y \
--out-bam /outputdir/SRR7890824-WGS_FD_T.bam
pbrun fq2bam 命令执行读取比对、排序、重复标记和索引;在四个 NVIDIA Tesla T4 GPU 上运行大约需要 50 分钟。fq2bam 的输出是一个 BAM 文件、一个 BAI 索引和一个 BQSR 报告文件。这些文件将用作下一步 SNP/indel 检测器的输入。
Parabricks 当前支持 DeepVariant、HaplotypeCaller 和 Mutect2 变异检测器。不同的检测器使用略有不同的方法来检测变异,每种方法在灵敏度和特异性之间都有权衡。此示例将运行 pbrun mutectcaller (Mutect2) 以生成一组小变异调用
$ docker run \
--gpus all \
--workdir /workdir \
--rm \
--volume $(pwd):/workdir \
--volume $(pwd):/outputdir \
nvcr.io/nvidia/clara/clara-parabricks:4.4.0-1 \
pbrun mutectcaller \
--ref /workdir/GRCh38.d1.vd1.fa \
--in-tumor-bam /workdir/SRR7890824-WGS_FD_T.bam \
--in-normal-bam /workdir/SRR7890827-WGS_FD_N.bam \
--in-tumor-recal-file /workdir/RR7890824-WGS_FD_T_BQSR_REPORT.txt \
--in-normal-recal-file /workdir/SRR7890827-WGS_FD_N_BQSR_REPORT.txt \
--out-vcf /outputdir/SRR7890824-SRR7890827-WGS_FD.vcf \
--tumor-name sm_SRR7890824 \
--normal-name sm_SRR7890827
此命令将调用的变异放入 SRR7890824-SRR7890827-WGS_FD.vcf 中,并创建 SRR7890824-SRR7890827-WGS_FD.vcf.stats 文件,该文件将在后续步骤中使用。
如果您正在寻找具有临床意义的变异,您可能会考虑运行其他变异检测器,并取两个或多个检测器的交集,以生成高度特异性的调用集。另一方面,如果您不太关心假阳性,但需要高度灵敏的调用集,则可以取多个调用器的并集。这种基于投票的共识方法已在许多大型研究中采用,以解决单个调用器的缺点。
首先运行 MuTect2,这是 Broad 开发的最广泛使用的变异检测器。Parabricks 提供了 MuTect2 v4.2.0.0 的加速版本,速度比社区版本快 10-15 倍。MuTect2 在四 T4 GPU 系统上应在 30 分钟内运行完毕。
MuTect2 的结果是纯文本 VCF 文件。
接下来,处理 Mutect2 VCF 文件,以使用 GATK4 SelectVariants 工具提取非插入缺失变异,该工具可以根据各种标准选择变异子集,以便于某些分析。此示例分析仅提取 SNV 和 MNV 变异,排除小插入缺失,以便进一步下游验证变异调用(与真值集比较)(请参阅下面的详细说明)
$ java -jar gatk-4.2.0.0/gatk-package-4.2.0.0-local.jar FilterMutectCalls \
-O SRR7890824-SRR7890827-WGS_FD.FilterMutectCalls.vcf \
-R GRCh38.d1.vd1.fa \
-V SRR7890824-SRR7890827-WGS_FD.vcf
获得 SNV/MNV .vcf 文件后,使用 GATK4 FilterMutectCalls 将过滤器应用于 Mutect2 的原始输出。M2FiltersArgumentCollection 中包含的参数在此处描述:here。
以下示例使用高置信度区域 bed 文件来与高置信度区域中的 Mutect2 变异调用相交,并使用 PASS 过滤器变异进行验证。
$ java -jar gatk-4.1.0.0/gatk-package-4.1.0.0-local.jar SelectVariants \
-R GRCh38.d1.vd1.fa \
-V SRR7890824-SRR7890827-WGS_FD.FilterMutectCalls.vcf \
--select-type-to-exclude INDEL \
-O SRR7890824-SRR7890827-WGS_FD.SNV-MNV.FilterMutectCalls.vcf
$ bedtools intersect \
-header \
-a SRR7890824-SRR7890827-WGS_FD.SNV-MNV.FilterMutectCalls.vcf \
-b High-Confidence_Regions_v1.2.bed > SRR7890824-SRR7890827-WGS_FD.SNV-MNV.FilterMutectCalls.hc.vcf
$ grep "#" SRR7890824-SRR7890827-WGS_FD.SNV-MNV.FilterMutectCalls.hc.vcf > mutect_header.txt
$ grep -v "#" SRR7890824-SRR7890827-WGS_FD.SNV-MNV.FilterMutectCalls.hc.vcf | awk '{if ($7 == "PASS")print}' > mutect_body.txt
$ cat mutect_header.txt mutect_body.txt > SRR7890824-SRR7890827-WGS_FD.SNV-MNV.FilterMutectCalls.hc.PASS.vcf
获得过滤后的 Mutect2 调用后,使用 R vcfR 和 upSetR 包生成 upset 图,以评估变异调用流程的性能。使用以下代码生成 upset 图
> library(vcfR)
> library(UpSetR)
> ## SEQC2 Somatic SNV upset plot
> ## Load the VCF files from truthset and Mutect2
> Truthset_snv_hc <- read.vcfR("high-confidence_sSNV_in_HC_regions_v1.2.hc.vcf.gz")
> mutect_snv_hc <- read.vcfR("SRR7890824-SRR7890827-WGS_FD.SNV-MNV.FilterMutectCalls.hc.PASS.vcf")
> ### extract chromosome and position to compare
> Truthset_snv_hc <-
as.vector(paste(Truthset_snv_hc@fix[, "CHROM"], Truthset_snv_hc@fix[, "POS"], sep = "_"))
> mutect_snv_hc <-
as.vector(paste(mutect_snv_hc@fix[, "CHROM"], mutect_snv_hc@fix[, "POS"], sep = "_"))
> ## create the read set
> read_sets <- list(Truthset_SEQC2_SNV = Truthset_snv_hc,
Mutect2 = mutect_snv_hc)
> upset(fromList(read_sets), order.by = c("freq"), keep.order = TRUE, sets = c("Mutect2", "Truthset_SEQC2_SNV"), point.size = 3.5, line.size = 2, mainbar.y.label = "Variant Intersections", text.scale = c(2, 2, 1.5, 1.5, 1.5, 1.5),sets.x.label = "Variants Calls Per Subset")
您应该得到类似于以下的图
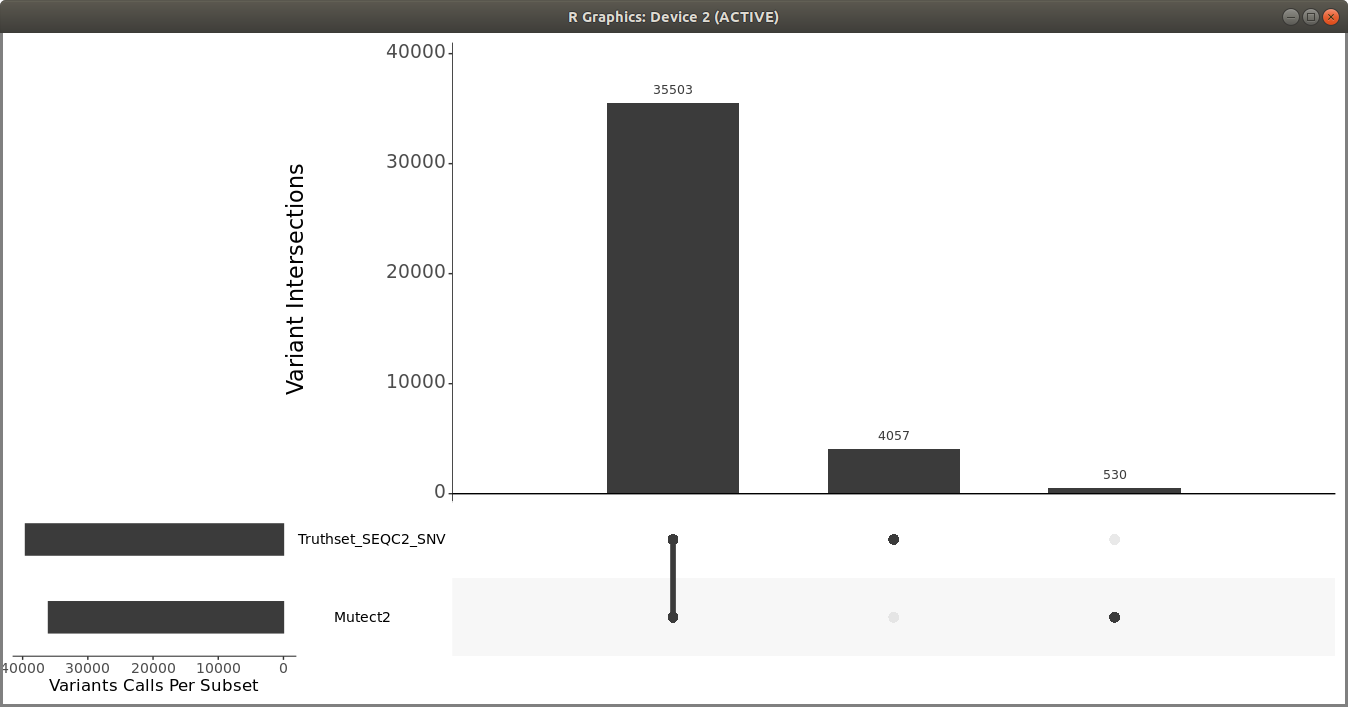
您还可以使用 Illumina hap.py 包中的 som.py 来比较两个 VCF 文件。
https://www.nvidia.com/en-us/clara/genomics/
https://sites.google.com/view/seqc2/home
https://gatk.broadinstitute.org/hc/en-us/articles/360036360312-Mutect2
https://github.com/Illumina/strelka/blob/v2.9.x/docs/userGuide/README.md
https://bioinformatics.mdanderson.org/public-software/muse/
https://csb5.github.io/lofreq/
https://github.com/genome/somatic-sniper/blob/master/gmt/documentation.md
https://github.com/Illumina/manta
https://github.com/Illumina/hap.py
https://cran.r-project.cn/web/packages/vcfR/readme/README.html
https://cran.r-project.cn/web/packages/UpSetR/vignettes/basic.usage.html