2. 交互式工作负载示例#
在本节中,我们将给出在您的 DGX Cloud 集群上的 Run:ai 上运行多个工作负载的说明。这些示例并非详尽无遗,但可以根据您自己的工作负载进行调整。
2.1. 交互式 NeMo 工作负载作业#
在本示例中,我们将逐步介绍如何使用 NGC 中的 NeMo 容器创建交互式工作负载。在 Run:ai 中,交互式工作负载称为工作空间。在这个特定的例子中,我们将运行一个 Jupyter notebook,使用 LoRA 对 LLM(Llama3-8B Instruct)针对 PubMedQA 数据集进行微调。
2.1.1. 先决条件和要求#
运行交互式 NeMo 作业之前,需要满足以下条件
您必须接受 NGC 组织的邀请,并将您的 NGC 凭据添加到 Run:ai。请在此处查看详细信息。
您必须具有用户角色L1 researcher、ML Engineer或系统管理员才能完成本教程的所有部分。
您的用户必须能够访问项目和部门。
至少一个具有 80 GB 内存的 A100 或更新的 GPU。
您必须能够访问一个计算资源,该资源在您的范围内至少创建了一个 GPU,您可以使用它。
您必须创建一个 Hugging Face 帐户,并同意 Meta Llama 3 社区许可证协议,同时登录您的 Hugging Face 帐户。然后,您必须在您的 帐户设置中生成 Hugging Face 读取访问令牌。访问 Jupyter Notebook 中的 Llama3 模型需要此令牌。
2.1.2. 创建数据源#
为了更轻松地在将来的作业中重用代码和检查点,创建了持久卷链 (PVC) 作为数据源。PVC 可以挂载在作业中,并在作业完成后持久存在,从而允许重用任何生成的数据。
要创建新的 PVC,请转到 数据源 页面。单击 新建数据源,然后单击 PVC 以打开 PVC 创建表单。
在新表单上,设置所需的范围。
重要提示
在集群或部门级别创建的 PVC 数据源 不会在项目或命名空间之间复制数据。每个项目或命名空间都将配置为单独的 PVC 副本,具有不同的底层 PV;因此,每个 PVC 中的数据不会被复制。
为 PVC 提供一个易于记忆的名称,如
nemo-lora-checkpoints,并根据需要添加描述。对于数据选项,请根据 PVC 建议 此处 选择适合您需求的新 PVC 存储类。在本示例中,
zonal-rwx已足够。要允许所有节点从 PVC 读取和写入,请为访问模式选择 多节点读写。输入10 TB作为大小,以确保我们有足够的容量用于将来的作业。选择 文件系统 作为卷模式。最后,将容器路径设置为/checkpoints,PVC 将在此处挂载在容器内部。完成后的部分应如下所示。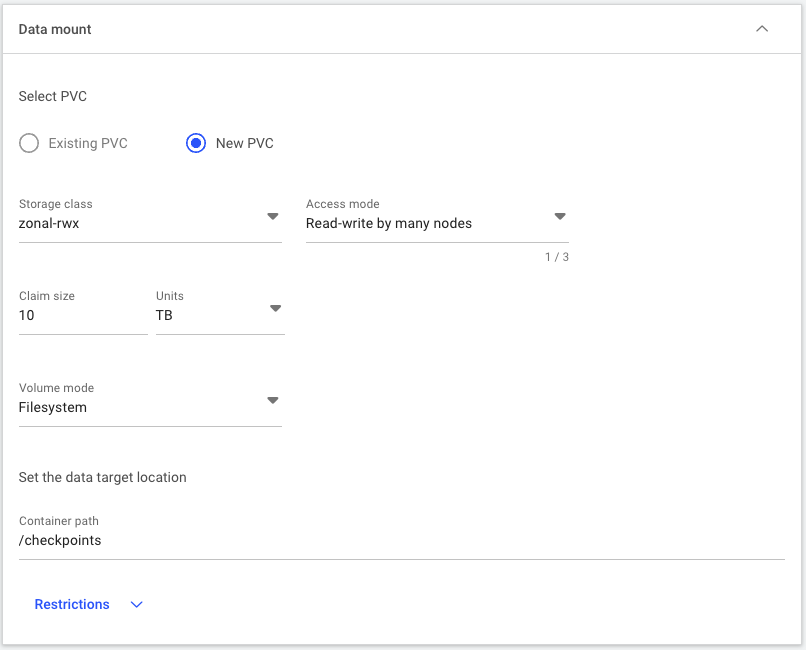
表单完成后,使用页面底部的按钮创建数据源。
注意
首次使用新 PVC 启动作业时,启动时间会更长,因为存储会在首次声明 PVC 时才进行配置。
2.1.3. 创建工作空间#
创建 PVC 后,我们可以配置工作空间,以告知 Run:ai 作业应如何启动。为此,请转到 工作负载 概览页面,然后单击左上角的 + 新建工作负载 按钮。将出现一个下拉菜单。从下拉菜单中,选择 工作空间。您将被带到 新建工作空间 创建页面。
选择要在其中运行作业的所需项目。
将表单的 模板 面板设置为 从头开始。
为您的工作空间输入一个描述性名称,例如
nemo-pubmedqa-lora。单击 继续。几秒钟后,将出现创建过程的 环境 面板。环境是一个模板,其中包含可重用于类似作业的常用设置。例如,环境可能基于 PyTorch 容器,并将 Jupyter 作为工具公开,使任何需要在 Jupyter notebook 中运行交互式 PyTorch 作业的人都可以使用相同的模板,而不是启动唯一的作业并在每次想要实验时重新配置设置。在本示例中,将基于 NeMo 容器创建一个新环境,并将 Jupyter 作为工具公开,以便将来更轻松地重用该模板。
要为 NeMo 作业创建新环境,请单击 环境 面板右上角的 + 新建环境 按钮。将打开 环境创建 表单。
在环境创建表单中,输入环境的名称,例如 “nemo-jupyter”,并可选择添加描述。
在 镜像 URL 下,输入
nvcr.io/nvidia/nemo:24.12。这将从 NGC 拉取 NeMo 容器。如果需要,可以更改镜像拉取策略。对于大多数情况,建议使用默认值“如果本地不存在”,但如果您要将新容器推送到具有相同标签的 NGC 组织,则应使用“始终从注册表拉取镜像”来检查镜像是否有更新。在 工作负载架构和类型 面板下,选择 标准 和 工作空间(如果尚未选择)。这些选项可能已选中并灰显。
单击 工具 面板以打开工具菜单,然后单击 +工具。此列表允许您向工作空间添加其他工具,以便更轻松地连接到其资源,例如用于连接到外部 Jupyter lab 会话的 Jupyter、用于连接到作业内部的 VS Code 服务器的 VS Code 等。
由于我们要运行交互式 Jupyter notebook,请在 选择工具 列表中选择 Jupyter。连接类型 下拉列表应显示 外部 URL、自动生成 和容器端口 “8888”。
单击 运行时设置 面板,然后单击以展开 命令和参数 面板。此部分允许我们根据需要覆盖默认容器运行设置。由于我们要启动 Jupyter lab 会话,请输入
jupyter-lab作为命令,以及--NotebookApp.base_url=/${RUNAI_PROJECT}/${RUNAI_JOB_NAME} --NotebookApp.token='' --ServerApp.allow_remote_access=true --allow-root --port=8888 --no-browser作为参数。这告诉 Run:ai 如何连接到 UI 中的 Jupyter lab 会话。对于目录,指定
/opt/NeMo作为在其中启动作业的目录。这将启动容器内/opt/NeMo目录中的 Jupyter lab 会话。单击页面右下角的 创建环境。您应该会看到一个弹出窗口,提示您的环境已创建。页面将刷新,您将被带回到 新建工作空间 创建页面的 环境 面板顶部。您刚刚创建的环境现在应已选中。
转到 计算资源 面板,为您的环境选择单 GPU 计算资源。虽然可以使用更多 GPU 进行 LoRA,但大多数任务只需一个 Ampere 或更新的 GPU,具有 80GB 内存,即可在相对较短的时间内完成。
在 数据源 部分中,选择您的
nemo-lora-checkpointsPVC。转到页面底部,然后单击 创建工作空间。这将把您的作业添加到队列中,一旦集群资源可用,它将自动被调度。
创建工作空间后,您将被带到工作负载概览页面,您可以在其中查看工作负载的状态。一旦状态显示为“运行中”,您的交互式工作负载就已准备就绪。如果您选择的容器以前从未被拉取到集群,并且 PVC 需要首次配置,则这可能需要一段时间。
一旦状态为“运行中”,您可以通过选中工作负载旁边的复选框,然后单击顶部菜单栏上的 连接 → Jupyter 来启动 Jupyter lab 会话。Jupyter lab 将在新窗口中打开。
2.1.4. 运行 Notebook#
连接到 Jupyter lab 会话后,通过打开容器自动打开的 /opt/NeMo 文件夹中的 tutorials/llm/llama-3 目录来导航到示例 notebook。此目录中有几个 notebook,包括 llama3-lora-nemofw.ipynb,这是我们将要遵循的 notebook。双击该文件以在主屏幕上打开 notebook。
该 notebook 逐步介绍了每个步骤的作用,但从高层次来看,它执行以下操作
从 Hugging Face 下载 Llama3-8B Instruct 模型
将模型转换为 .nemo 格式,以便 NeMo 工具包轻松摄取
下载和预处理 PubMedQA 数据集
使用 LoRA PEFT 技术针对 PubMedQA 数据集微调 Llama3-8B Instruct 模型
部署模型以进行本地推理
针对测试数据集评估模型,以确定微调模型的总体准确性
可以逐个单元格逐步执行 notebook,也可以一次全部运行,并在以后查看结果。唯一重要的注意事项是,单元格 2 要求您添加 Hugging Face 访问令牌,如单元格文本的要求部分所列,以便通过 Hugging Face 身份验证以下载模型。
随着 notebook 的运行,您可以导航回 Run:ai 工作负载页面,您的作业现在会显示基于资源使用情况的指标。“指标”选项卡显示作业期间的 GPU 和 CPU 计算和内存使用情况。这是确定作业性能的绝佳资源。
2.1.5. 将模型持久化到存储#
由于 PVC 已添加到工作空间并挂载在容器内的 /checkpoints,因此保存到容器内 /checkpoints 的任何文件或目录都将保存在 PVC 中,并在将来的作业中可用。
要将生成的模型保存到 PVC,请在 Jupyter lab 会话中打开一个终端选项卡,并使用以下命令导航到 notebook 目录
cd /opt/NeMo/tutorials/llm/llama-3
该模型保存在
results/Meta-Llama-3-8B-Instruct/checkpoints/megatron_gpt_peft_lora_tuning.nemo。可以使用以下命令进行验证$ du -sh results/Meta-Llama-3-8B-Instruct/checkpoints/megatron_gpt_peft_lora_tuning.nemo 21M results/Meta-Llama-3-8B-Instruct/checkpoints/megatron_gpt_peft_lora_tuning.nemo
要将文件复制到 PVC,请运行
cp results/Meta-Llama-3-8B-Instruct/checkpoints/megatron_gpt_peft_lora_tuning.nemo /checkpoints
一旦检查点被复制,它将在所有使用同一 PVC 的未来工作负载中可用,除非该文件被有意删除。
2.1.6. 清理环境#
作业完成后,可以将其删除以释放资源供其他作业使用。请注意,某些作业不会自行终止,应进行监控以确保已完成作业的资源不会闲置。
2.2. RAPIDS 和 Polars 工作空间#
在本示例中,我们将逐步介绍创建另一个工作空间的过程,该工作空间使用 NGC 中的 RAPIDS 容器。我们将启动一个 Jupyter 环境来试验 RAPIDS 和 Polars 之间 新的集成。
2.2.1. 先决条件和要求#
运行 RAPIDS 工作空间之前,需要满足以下条件
您必须具有用户角色L1 researcher、ML Engineer或系统管理员才能完成本教程的所有部分。
您的用户必须能够访问项目和部门。
您的 DGX Cloud 集群中至少有一个空闲 GPU。
您必须能够访问一个计算资源,该资源在您的范围内至少创建了一个 GPU,您可以使用它。
2.2.2. 创建工作空间#
转到 工作负载 概览页面,然后单击左上角的 + 新建工作负载 按钮。将出现一个下拉菜单。从下拉菜单中,选择 工作空间。您将被带到 新建工作空间 创建页面。
选择要在其中运行作业的所需项目。
将表单的 模板 面板设置为 从头开始。
为您的工作空间输入一个描述性名称,例如
rapids-polars。单击 继续。几秒钟后,将出现创建过程的 环境 面板。要为 RAPIDS 工作空间创建新环境,请单击 环境 面板右上角的 + 新建环境 按钮。将打开 环境创建 表单。
在环境创建表单中,输入环境的名称,例如 “rapids-jupyter”,并可选择添加描述。
在 镜像 URL 下,输入
nvcr.io/nvidia/rapidsai/notebooks:24.08-cuda11.8-py3.9。如果需要,可以更改镜像拉取策略。对于大多数情况,建议使用默认值“如果本地不存在”,但如果您要将新容器推送到具有相同标签的 NGC 组织,则应使用“始终从注册表拉取镜像”来检查镜像是否有更新。在 工作负载架构和类型 面板下,选择 标准 和 工作空间(如果尚未选择)。这些选项可能已选中并灰显。
单击 工具 面板以打开工具菜单,然后单击 +工具。此列表允许您向工作空间添加其他工具,以便更轻松地连接到其资源,例如用于连接到外部 Jupyter lab 会话的 Jupyter、用于连接到工作空间内部的 VS Code 服务器的 VS Code 等。
由于我们要运行 Jupyter notebook,请在 选择工具 列表中选择 Jupyter。连接类型 下拉列表应显示 外部 URL、自动生成 和容器端口 “8888”。
单击 运行时设置 面板,然后单击以展开 命令和参数 面板。此部分允许我们根据需要覆盖默认容器运行设置。由于我们要启动 Jupyter lab 会话,请输入
jupyter-lab作为命令,以及--NotebookApp.base_url=/${RUNAI_PROJECT}/${RUNAI_JOB_NAME} --NotebookApp.token='' --ServerApp.allow_remote_access=true --allow-root --port=8888 --no-browser作为参数。这告诉 Run:ai 如何连接到 UI 中的 Jupyter lab 会话。不要修改任何其他设置。
单击页面右下角的 创建环境。您应该会看到一个弹出窗口,提示您的环境已创建。页面将刷新,您将被带回到 新建工作空间 创建页面的 环境 面板顶部。您刚刚创建的环境现在应已选中。
转到 计算资源 面板,为您的环境选择单 GPU 计算资源。
转到页面底部,然后单击 创建工作空间。这将把您的作业添加到队列中,一旦集群资源可用,它将自动被调度。
创建工作空间后,您将被带到工作负载概览页面,您可以在其中查看工作负载的状态。一旦状态显示为“运行中”,您的工作空间就已准备就绪。如果您选择的容器以前从未被拉取到集群,则这可能需要一段时间。
一旦状态为“运行中”,您可以通过选中工作负载旁边的复选框,然后单击顶部菜单栏上的 连接 → Jupyter 来启动 Jupyter lab 会话。Jupyter lab 将在新窗口中打开。
2.2.3. 下载 Polars Notebook#
连接到 Jupyter lab 会话后,您可以导航到 notebooks 目录中的集成 RAPIDS notebook 示例,并在您的工作空间中进行实验。
要开始下载 GPU 加速的 Polars 示例 notebook,请单击 终端 框打开终端界面。
在生成的终端中,运行以下命令以下载新的 notebook。
wget https://raw.githubusercontent.com/rapidsai-community/showcase/refs/heads/main/accelerated_data_processing_examples/polars_gpu_engine_demo.ipynb
2.2.4. 运行 Polars Notebook#
该 notebook 介绍了 Polars 是什么以及最适合在何处使用,以及以下代码
安装带有 GPU 引擎的 Polars,以及其他 notebook 依赖项
下载示例 Kaggle 数据集
初始化 Polars 以在 DGX Cloud 中使用 GPU
运行各种示例分析,以提供仅使用 CPU 的 Polars 和使用 GPU 的 Polars 之间的性能比较
要打开 notebook,请在 polars_gpu_engine_demo.ipynb 文件在 Jupyter 文件导航窗格中变为可见时双击它。
可以逐个单元格逐步执行 notebook,也可以一次全部运行,并在以后查看结果。
随着 notebook 的运行,您可以导航回 Run:ai 工作负载页面,您的作业现在会显示基于资源使用情况的指标。“指标”选项卡显示作业期间的 GPU 和 CPU 计算和内存使用情况。这是确定作业性能的绝佳资源。在您正在使用的 Jupyter notebook 页面中,您还可以使用 RAPIDS NGC 容器中的集成 GPU 性能可视化插件(它是页面左侧带有 GPU 图标的选项卡)。
2.2.5. 清理环境#
完成工作负载后,可以停止或删除它,以释放资源供其他工作负载使用。请注意,某些工作负载不会自行终止,应监控工作负载以确保已完成工作负载的资源不会闲置。
2.3. 在工作负载内部运行 Visual Studio Code#
在本示例中,我们将逐步介绍如何通过 Run:ai 命令行界面 (CLI) 创建一个工作空间,该工作空间运行 VS Code 实例,并将必要的端口公开到本地工作站,以便访问 VS Code Web 界面。
2.3.1. 先决条件和要求#
运行 VS Code 工作空间之前,需要满足以下条件
您必须具有用户角色L1 researcher、ML Engineer或系统管理员才能完成本教程的所有部分。
您的用户必须能够访问项目和部门。
您必须已安装 Run:ai CLI 工具,如 高级文档 中所述。
2.3.2. 通过本地工作站上的 CLI 创建工作空间#
在您的工作站(或其他已安装和配置 Run:ai CLI 的系统)上打开终端。
注意
如果您安装 Run:ai CLI 的系统是远程系统,您可以 SSH 到该系统并创建本地端口转发,将 Run:ai 将使用的端口与本地工作站上的同一端口连接起来。例如,
ssh -L 8080:localhost:8080 the-ip-or-hostname-of-your-remote-system。记下您的目标项目。
在终端中输入以下命令和参数(将您的目标项目作为
--project标志的参数插入),然后按 Enter 键。runai submit vscode --project your-project-name-here --image linuxserver/code-server:4.92.2 --environment PUID=0 --environment PGID=0 --interactive
(可选)使用以下命令验证项目是否正在运行。
runai describe job vscode --project your-project-name-here
通过 Run:ai CLI 将端口转发到您的本地工作站,使用以下命令。参数
port中的第一个端口是任意的 - 它可以更改为您系统上任何未使用的端口,而不是8080。runai port-forward vscode --project your-project-name-here --port 8080:8443
要访问 VS Code Web 界面,请导航到 https://:8080。
2.3.3. 清理环境#
完成工作负载后,可以停止或删除它,以释放资源供其他工作负载使用。要从 CLI 删除工作负载,请使用以下命令。
runai delete --project demo-project job vscode
2.4. 将 WandB 与工作空间一起使用#
在本示例中,我们将创建一个交互式工作负载,其中包含用于实验跟踪的 Weights and Biases (WandB) 集成。对于训练工作负载,可以按照与创建和添加凭据和环境变量类似的过程进行操作。一旦 API 密钥连接到您的工作负载,您的 WandB 运行应自动连接到您的帐户。
2.4.1. 先决条件和要求#
将 WandB 集成与交互式工作负载一起使用之前,需要满足以下条件
您必须具有 L1 researcher、ML Engineer 或 系统管理员 的用户角色才能完成本教程的所有部分。
您的用户必须能够访问项目和部门。
您必须能够访问您范围内的计算资源,以便使用。
您必须拥有 Weights and Biases 帐户并拥有 API 密钥。
2.4.2. 创建凭据#
为了安全地将必要的密钥插入到工作负载中,我们将为访问密钥 ID 和秘密访问密钥创建一个 凭据,从而允许访问 WandB。
要创建新的凭据,请转到 凭据 页面。单击 + 新建凭据,然后单击 访问密钥 以打开访问密钥创建表单。
在该表单上,设置所需的范围。范围应尽可能窄 - 在本例中,将范围设置为与您的工作负载的目标项目匹配。
为访问密钥提供一个描述性名称,例如
wandb-access-key,并根据需要添加描述。在 密钥 面板中,选择 新建密钥。将 访问密钥 字段设置为
WANDB_API_KEY,并将 访问密钥 字段设置为您的 WandB API 密钥(可以在 WandB 站点上的个人资料设置中找到)。单击 创建凭据 以保存新的凭据。
2.4.3. 创建工作空间#
转到 工作负载 概览页面,然后单击左上角的 + 新建工作负载 按钮。将出现一个下拉菜单。从下拉菜单中,选择 工作空间。您将被带到 新建工作空间 创建页面。
选择要在其中运行作业的所需项目。
将表单的 模板 面板设置为 从头开始。
为您的工作负载输入一个描述性名称,例如
wandb-training。单击 继续。几秒钟后,将出现创建过程的 环境 面板。要为工作负载创建新环境,请单击 环境 面板右上角的 + 新建环境 按钮。将打开 环境创建 表单。
在 环境创建 表单中,输入环境的名称,例如
wandb-training-env,并可选择添加描述。在 镜像 URL 下,输入
nvcr.io/nvidia/pytorch:24.08-py3。如果需要,可以更改镜像拉取策略。对于大多数情况,建议使用默认值“如果本地不存在”,但如果您要将新容器推送到具有相同标签的 NGC 组织,则应使用“始终从注册表拉取镜像”来检查镜像是否有更新。在 工作负载架构和类型 面板下,选择 标准 和 工作空间(如果尚未选择)。这些选项可能已选中并灰显。
单击 工具 面板以打开工具菜单,然后单击 +工具。此列表允许您向工作空间添加其他工具,以便更轻松地连接到其资源,例如用于连接到外部 Jupyter lab 会话的 Jupyter、用于连接到工作空间内部的 VS Code 服务器的 VS Code 等。
由于我们要运行 Jupyter notebook,请在 选择工具 列表中选择 Jupyter。连接类型 下拉列表应显示 外部 URL、自动生成 和容器端口 “8888”。
单击 运行时设置 面板,然后单击以展开 命令和参数 面板。此部分允许我们根据需要覆盖默认容器运行设置。由于我们要启动 Jupyter lab 会话,请输入
jupyter-lab作为命令,以及--NotebookApp.base_url=/${RUNAI_PROJECT}/${RUNAI_JOB_NAME} --NotebookApp.token='' --ServerApp.allow_remote_access=true --allow-root --port=8888 --no-browser作为参数。这告诉 Run:ai 如何连接到 UI 中的 Jupyter lab 会话。不要修改任何其他设置。
单击页面右下角的 创建环境。您应该会看到一个弹出窗口,提示您的环境已创建。页面将刷新,您将被带回到 新建工作空间 创建页面的 环境 面板顶部。您刚刚创建的环境现在应已选中。
转到 计算资源 面板,为您的环境选择单 GPU 计算资源。
要将 凭据 嵌入到工作负载中,请单击 + 环境变量 按钮。
对于 名称 字段,输入
WANDB_API_KEY,将 来源 设置为凭据,将 凭据名称 字段设置为wandb-access-key,并将 密钥 字段设置为SecretKey。注意
如果您的 WandB 项目连接到特定的 WandB 团队,则可能需要额外的环境变量。对于 名称 字段,输入
WANDB_ENTITY,将 来源 设置为自定义,并将 值 字段设置为您的团队名称。转到 计算资源 面板,然后选择您所需的计算资源。对于本示例,单个 GPU 资源就足够了。
转到页面底部,然后单击 创建工作空间。这将把您的工作负载添加到队列中,一旦集群资源可用,它将自动被调度。
创建工作空间后,您将被带到工作负载概览页面,您可以在其中查看工作负载的状态。一旦状态显示为“运行中”,您的工作空间就已准备就绪。如果您选择的容器以前从未被拉取到集群,则这可能需要一段时间。
一旦状态为“运行中”,您可以通过选中工作负载旁边的复选框,然后单击顶部菜单栏上的 连接 → Jupyter 来启动 Jupyter lab 会话。Jupyter lab 将在新窗口中打开。
2.4.4. 在 Jupyter Notebook 中连接到 WandB#
PyTorch 没有在容器中自动集成 WandB。在您的 Jupyter lab 会话中打开一个终端选项卡以下载 WandB 客户端。运行以下命令
pip install wandb
打开一个新的 notebook 并运行以下代码以测试您的 WandB 连接
import wandb import random # start a new wandb run to track this script wandb.init( # set the wandb project where this run will be logged project="my-awesome-project", # track hyperparameters and run metadata config={ "learning_rate": 0.02, "architecture": "CNN", "dataset": "CIFAR-100", "epochs": 10, } ) # simulate training epochs = 10 offset = random.random() / 5 for epoch in range(2, epochs): acc = 1 - 2 ** -epoch - random.random() / epoch - offset loss = 2 ** -epoch + random.random() / epoch + offset # log metrics to wandb wandb.log({"acc": acc, "loss": loss}) # [optional] finish the wandb run, necessary in notebooks wandb.finish()
运行此代码后,您应该在您的 WandB 帐户中看到一个名为
my-awesome-project的新项目。
对于您自己的 WandB 实验,添加 API 密钥将自动执行登录过程,因此您自己的代码应自动连接到您的帐户运行。
2.4.5. 清理环境#
工作负载完成后,可以从 工作负载 页面删除它,或将其保留作为将来数据下载工作负载的参考。
2.5. 使用 BioNeMo 框架进行 ESM-2nv 数据预处理和模型训练#
本教程提供了一个使用 BioNeMo 框架训练 BioNeMo 大型语言模型的用例示例。本教程将帮助用户体验
为 ESM-2nv 预处理 UniRef50 和 UniRef90 数据
预训练和从检查点继续训练 ESM-2nv
在 DGX Cloud 上的 Run:ai 上启动交互式和训练工作负载
有关 NVIDIA BioNeMo 框架和 ESM-2nv 模型的更多信息,请参阅 BioNeMo 文档。
2.5.1. 先决条件和要求#
运行交互式 NeMo 作业之前,需要满足以下条件
您必须接受 NGC 组织的邀请,并将您的 NGC 凭据添加到 Run:ai。请参阅 访问您的 NGC 组织。
您必须具有 L1 researcher、ML Engineer 或 系统管理员 的用户角色才能完成本教程的所有部分。
您的用户必须能够访问项目和部门。
至少一个具有 80 GB 内存的 A100 或更新的 GPU。
您必须能够访问一个计算资源,该资源在您的范围内至少创建了一个 GPU,您可以使用它。
2.5.2. 创建数据源#
我们将创建一个 PVC 数据源,以便与未来的工作负载共享结果数据集。
要创建新的 PVC,请转到 数据源 页面。单击 新建数据源,然后单击 PVC 以打开 PVC 创建表单。
在新表单上,设置所需的范围。
重要提示
在集群或部门级别创建的 PVC 数据源 不会在项目或命名空间之间复制数据。每个项目或命名空间都将配置为单独的 PVC 副本,具有不同的底层 PV;因此,每个 PVC 中的数据不会被复制。
为 PVC 提供一个易于记忆的名称,如
ems2-workspace,并根据需要添加描述。对于数据选项,根据PVC 推荐存储类,选择适合您需求的新 PVC 存储类。在本例中,
zonal-rwx就足够了。要允许所有节点从 PVC 读取和写入,请为访问模式选择多节点读写。输入适合您的目标数据集的容量——在本例中,我们将指定10 TB。选择 Filesystem 作为卷模式。最后,将容器路径设置为/esm2-workspace,PVC 将挂载到容器内。表单完成后,单击页面底部的 CREATE DATA SOURCE(创建数据源)按钮。
注意
首次使用新 PVC 启动作业时,启动时间会更长,因为存储会在首次声明 PVC 时才进行配置。
2.5.3. 创建用于预处理的交互式工作空间#
创建 PVC 后,我们可以配置工作空间,以告知 Run:ai 作业应如何启动。为此,请转到 工作负载 概览页面,然后单击左上角的 + 新建工作负载 按钮。将出现一个下拉菜单。从下拉菜单中,选择 工作空间。您将被带到 新建工作空间 创建页面。
选择要在其中运行作业的所需项目。
将表单的 模板 面板设置为 从头开始。
为您的工作空间输入一个描述性名称,例如
esm2-preprocessing。单击 Continue(继续)。几秒钟后,将出现创建过程的 Environment(环境)窗格。环境是一个模板,其中包含可重用于类似作业的常用设置。例如,环境可能基于 PyTorch 容器,并将 Jupyter 作为工具公开,使任何需要在 Jupyter notebook 中运行交互式 PyTorch 作业的人都可以使用相同的模板,而不是启动唯一的作业并在每次想要实验时重新配置设置。在本示例中,将基于 NeMo 容器创建一个新环境,并将 Jupyter 作为工具公开,以便将来更轻松地重用该模板。
要为 BioNeMo 作业创建一个新环境,请单击 Environment(环境)窗格右上角的 + New Environment(+ 新环境)按钮。将打开 Environment creation(环境创建)表单。
在 Environment creation(环境创建)表单中,输入环境的名称,例如“bionemo-jupyter”,并可选择添加描述。
在 Image URL(镜像 URL)下,输入
nvcr.io/nvidia/clara/bionemo-framework:1.9。这将从 NGC 拉取 BioNeMo 容器。注意
如果需要,可以更改镜像拉取策略。建议大多数情况下使用默认值“if not already present”(如果尚不存在),但如果您要将新容器使用相同的标签推送到 NGC 组织,则应使用“always pull the image from the registry”(始终从注册表拉取镜像)来检查镜像的更新。
在 工作负载架构和类型 面板下,选择 标准 和 工作空间(如果尚未选择)。这些选项可能已选中并灰显。
单击 工具 面板以打开工具菜单,然后单击 +工具。此列表允许您向工作空间添加其他工具,以便更轻松地连接到其资源,例如用于连接到外部 Jupyter lab 会话的 Jupyter、用于连接到作业内部的 VS Code 服务器的 VS Code 等。
由于我们想要运行交互式 Jupyter notebook,请在 Select tool(选择工具)列表中选择 Jupyter。Connection type(连接类型)下拉列表应显示 External URL(外部 URL)、Auto generate(自动生成)和 Container port(容器端口)“8888”。
单击 运行时设置 面板,然后单击以展开 命令和参数 面板。此部分允许我们根据需要覆盖默认容器运行设置。由于我们要启动 Jupyter lab 会话,请输入
jupyter-lab作为命令,以及--NotebookApp.base_url=/${RUNAI_PROJECT}/${RUNAI_JOB_NAME} --NotebookApp.token='' --ServerApp.allow_remote_access=true --allow-root --port=8888 --no-browser作为参数。这告诉 Run:ai 如何连接到 UI 中的 Jupyter lab 会话。单击页面右下角的 创建环境。您应该会看到一个弹出窗口,提示您的环境已创建。页面将刷新,您将被带回到 新建工作空间 创建页面的 环境 面板顶部。您刚刚创建的环境现在应已选中。
转到 Compute resource(计算资源)窗格,为您的环境选择单 GPU 计算资源。
在 Data Sources(数据源)部分,选择您的
esm2-workspacePVC。转到页面底部,然后单击 创建工作空间。这将把您的作业添加到队列中,一旦集群资源可用,它将自动被调度。
创建工作空间后,您将被带到工作负载概览页面,您可以在其中查看工作负载的状态。一旦状态显示为“运行中”,您的交互式工作负载就已准备就绪。如果您选择的容器以前从未被拉取到集群,并且 PVC 需要首次配置,则这可能需要一段时间。
一旦状态为“运行中”,您可以通过选中工作负载旁边的复选框,然后单击顶部菜单栏上的 连接 → Jupyter 来启动 Jupyter lab 会话。Jupyter lab 将在新窗口中打开。
2.5.4. 使用工作空间预处理 ESM-2nv 的数据#
为了简要展示 BioNeMo 框架的模型训练能力,我们将使用 UniRef50 和 UniRef90 数据集来提供多样化且非冗余的蛋白质序列集。通过同时使用两者,模型可以从广泛的序列变异中学习,同时避免冗余。这有助于捕获与蛋白质功能和结构预测相关的多样化特征和模式,同时防止过拟合并提高泛化能力。出于演示目的,部分示例数据集位于 ${BIONEMO_HOME}/examples/tests/test_data/uniref202104_esm2_qc 中。
在您的 Jupyter lab 会话中打开一个终端选项卡。
数据存储在 zip 文件中,因此运行以下命令以将原始 FASTA 文件和集群映射文件提取到您的 PVC 中。您还将把您的 PVC 数据路径保存为
DATASET_DIR以供将来步骤使用。export ZIP_FILE=${BIONEMO_HOME}/examples/tests/test_data/uniref202104_esm2_qc_test200_val200.zip export DATASET_DIR=/esm2-workspace/test_data unzip $ZIP_FILE -d $DATASET_DIR/
mapping.tsv文件用于将蛋白质序列与其各自的集群关联起来。这有助于减少冗余、组织数据和评估模型性能,方法是跟踪序列相似性并确保多样化的训练数据。使用此文件的解压内容,我们首先创建预处理的
/train、/val和/test文件夹,将蛋白质序列组织成批处理 CSV 文件。如果您计划按最初创建的方式使用 ESM-2,则务必同时使用这两个数据集。同样的方法适用于集群映射文件。ESM2Preprocess 类可以间接地处理集群,作为数据集准备过程的一部分。它利用 UniRef50 到 UniRef90 的集群映射来组织蛋白质序列,确保数据被适当地集群用于训练和验证。
请注意,此脚本本身不执行集群操作,而是依赖于 TSV 文件格式中提供的预定义集群映射来组织蛋白质序列。预期的格式是一个 TSV 文件,其中第一列表示集群 ID(UniRef50 中的 FASTA 标头),第二列列出成员,成员之间用逗号分隔。这些成员对应于 UniRef90 FASTA 文件中的条目。
现在,我们可以运行预处理脚本。预处理步骤包括
从指定的 URL 或 NGC 注册表下载数据集。
如果需要,提取并解压缩下载的数据。
使用 pyfastx 索引 FASTA 文件,以方便数据访问。
将数据集拆分为训练集、验证集和测试集。
将 FASTA 序列转换为 CSV 格式,如果需要,将其划分为多个文件。
如果特定用例需要,则生成其他文件,如 memmap 或排序的 FASTA 文件。
有关预处理步骤的更多详细信息,请查阅
../bionemo/data/preprocess/protein/preprocess.py文件和此处的文档。要预处理数据,请使用 pretrain.py 脚本并将
do_training参数设置为False,如下所示。有关更多信息,请参阅 Command Line and YAML Configurations(命令行和 YAML 配置)。cd ${BIONEMO_HOME} export TRAIN_UF50_FASTA=/esm2-workspace/test_data/uniref202104_esm2_qc_test200_val200/uniref50_train_filt.fasta export TRAIN_UF90_FASTA=/esm2-workspace/test_data/uniref202104_esm2_qc_test200_val200/ur90_ur50_sampler.fasta export TRAIN_CLUSTER_MAPPING_TSV=/esm2-workspace/test_data/uniref202104_esm2_qc_test200_val200/mapping.tsv export DATAPATH=/esm2-workspace/test_data/uniref202104_esm2_qc_test200_val200 python examples/protein/esm2nv/pretrain.py \ --config-path=conf \ --config-name=pretrain_esm2_650M \ ++do_training=False \ ++do_preprocessing=True \ ++model.data.val_size=500 \ ++model.data.test_size=100 \ ++model.data.train.uf50_datapath=${TRAIN_UF50_FASTA} \ ++model.data.train.uf90_datapath=${TRAIN_UF90_FASTA} \ ++model.data.train.cluster_mapping_tsv=${TRAIN_CLUSTER_MAPPING_TSV} \ ++model.data.dataset_path=${DATAPATH}
命令行和 YAML 配置
以
--开头的参数作为命令行参数传递给pretrain.py。这些参数包括--config-path和--config-name这些指定配置的文件夹和 YAML 文件名。路径相对于
pretrain.py。例如config-path:指的是配置文件夹,例如 examples/protein/esm2nv/conf。
config-name:指的是 YAML 配置文件,例如 pretrain_esm2_650M.yaml。
本例中配置文件的完整路径将是
{BIONEMO_HOME}/examples/protein/esm2nv/conf/pretrain_esm2_650M.yaml以 ++ 开头的参数可在 YAML 文件中配置。以下是在
pretrain_esm2_650M.yaml文件中找到的此类参数的一些示例,该文件继承自 base_config.yamldo_training:如果您只想预处理数据而不启动训练,请设置为 False。
model.data.val_size和model.data.test_size这些分别指定验证数据集和测试数据集的大小。
model.data.train.uf50_datapath:指定 UniRef50 FASTA 文件的路径。
model.data.train.uf90_datapath:指定 UniRef90 FASTA 文件的路径。
model.data.train.cluster_mapping_tsv:指定映射文件的路径,该文件将 UniRef50 集群映射到 UniRef90 序列。
model.data.dataset_path:指定预处理的 UniRef50 和 UniRef90 数据的输出目录的路径。处理后,将创建以下目录
uf50:包含 train/test/val 拆分,每个拆分都包含类似
x000.csv的文件。uf90:包含名为 uf90_csvs 的文件夹,其中包含类似
x000.csv的文件。请注意,此目录中不会有 train/test/val 拆分,因为 UniRef90 仅在训练期间使用。
也可以直接对 YAML 文件进行更改,而不是通过命令行覆盖参数。
现在我们准备好在单个节点上进行预训练。对于多节点训练,请继续执行以下步骤。
对于多节点训练,我们需要在我们的 PVC 中创建一个运行脚本。我们将启动一个
run.sh脚本,它将使用torchrun运行pretrain.py脚本,torchrun 是一种运行多进程应用程序的工具,其中每个进程都分配了一个唯一的 rank。DGX Cloud 上提供的 PyTorch Training Operator 与 torchrun 协调工作,以根据为作业分配的 GPU 总数自动设置RANK、LOCAL_RANK和WORLD_SIZE以及其他环境变量。使用终端选项卡,在您的 PVC 中创建运行脚本。此代码将运行一个双节点分布式训练作业。要更改节点数,请修改++trainer.num_nodes配置。cat << EOF > /esm2-workspace/run.sh #!/bin/bash cd \${BIONEMO_HOME}/examples/protein/esm2nv/ && torchrun pretrain.py \ --config-path=conf \ --config-name=pretrain_esm2_650M do_training=True ++trainer.max_steps=1000 ++trainer.val_check_interval=100 \ ++trainer.devices=8 \ ++trainer.num_nodes=2 \ ++model.data.dataset_path=/esm2-workspace/test_data/uniref202104_esm2_qc_test200_val200 \ ++model.micro_batch_size=2 \ ++trainer.val_check_interval=12 \ ++exp_manager.create_wandb_logger=False \ ++exp_manager.exp_dir=/esm2-workspace/results EOF
注意
要从现有检查点继续训练,请使用
exp_manager.resume_if_exists=True。要使用 Weights and Biases 进行实验管理和训练可视化,请设置
++exp_manager.create_wandb_logger=True。创建工作负载时,还需要添加您的WANDB_API_KEY环境变量,以允许 WandB 登录。请参阅将 WandB 与工作空间一起使用。
2.5.5. 预训练#
现在,我们将使用我们准备好的数据和位于 ${BIONEMO_HOME}/examples/protein/esm2nv/conf 文件夹中的 pretrain_esm2_650M.yaml 配置文件中提供的参数,从头开始执行 ESM-2 的预训练。这可以在 Jupyter notebook 中完成,也可以使用分布式训练完成。
2.5.5.1. Jupyter Notebook 中的单 GPU 训练#
对于此演示示例,我们将通过设置以下参数来缩短训练所需的时间:++trainer.max_steps=1 和 ++val_check_interval=1。用户可以通过编辑 .yaml 配置文件或通过使用 Hydra 在运行时覆盖配置参数来更新这些参数,如下例所示。
python examples/protein/esm2nv/pretrain.py \
--config-path=conf \
--config-name=pretrain_esm2_650M \
name=esm2_pretrain \
++do_training=True \
++model.data.dataset_path=/esm2-workspace/test_data/uniref202104_esm2_qc_test200_val200 \
++exp_manager.exp_dir=/esm2-workspace \
++trainer.devices=1 \
++model.micro_batch_size=1 \
++trainer.max_steps=1 \
++trainer.val_check_interval=1 \
++exp_manager.create_wandb_logger=False \
++trainer.limit_train_batches=1 \
++trainer.limit_val_batches=1
如果您有硬件限制,可以将使用的配置更改为 pretrain_esm2_8M。请参阅下面 YAML 配置的说明。
YAML 配置
trainer.devices:指定每个节点要使用的 GPU 数量。
trainer.max_steps:设置最大训练步数。
trainer.val_check_interval:确定运行验证的频率。
trainer.limit_train_batches和trainer.limit_val_batches分别限制训练和验证的批次数。
model.micro_batch_size:指的是在执行权重更新之前,在单个前向/后向传递中每个 rank 处理的样本数。
2.5.5.2. 多节点分布式预训练#
要创建训练作业,请导航到 Workloads(工作负载)页面,然后单击 New Workload > Training(新建工作负载 > 训练)。
在项目创建表单页面上,选择要在其上运行作业的所需项目。
为工作负载架构选择 Distributed(分布式)以运行多节点作业。这将添加一个下拉菜单,其中列出了用于运行分布式作业的可用框架。由于我们正在运行 PyTorch 作业,请选择 PyTorch 框架。
对于分布式训练配置,可以选择 Workers & master(工作节点和主节点)或 Workers only(仅工作节点)。
注意
在本文档中,我们将使用术语“primary”(主节点)而不是“master”(主节点),以与现代术语保持一致。请注意,UI 和命令可能仍将其称为“master”(主节点)。
当节点需要相互协调时(例如在执行 all_reduce 操作的作业中),通常需要主节点。主节点可以与工作节点相同并运行训练过程,也可以是仅执行协调的轻量级 pod。如果作业不需要进程之间的协调,则可以使用 Workers only(仅工作节点)选项。对于我们的示例,我们希望 pod 相互协调。选择 Workers & master(工作节点和主节点)选项。
确保为模板选择 Start from scratch(从头开始)选项。
为作业指定一个名称,如
ems2nv-pretraining,然后单击 Continue(继续)。在新打开的环境表单中,选择 New environment(新建环境)。在新表单中,提供环境的名称,例如“distributed-bionemo-env”,并可选择添加描述。
在 Image URL(镜像 URL)下,输入
nvcr.io/nvidia/clara/bionemo-framework:1.9。这将从 NGC 拉取 BioNeMo 容器。如果需要,可以更改镜像拉取策略。建议大多数情况下使用默认值“if not already present”(如果尚不存在),但如果您要将新容器使用相同的标签推送到 NGC 组织,则应使用“always pull the image from the registry”(始终从注册表拉取镜像)来检查镜像的更新。表单上的大多数其余设置可以保留为默认值。我们将在工作节点中更改运行时设置,以保持环境的灵活性,供将来使用。
选择 Create Environment(创建环境)按钮后,您将返回到作业创建页面,并已选择新环境。
在 Commands and Arguments(命令和参数)字段中,输入命令
sh /esm2-workspace/run.sh。这将使用我们在预处理期间创建的运行脚本来运行分布式训练作业。在计算资源窗格中,选择所需的工作节点数。请注意,由于我们使用的是主节点和工作节点配置,并且我们希望主节点参与训练,因此我们将指定比作业预期的总节点数少一个的工作节点。换句话说,如果我们想要运行一个 8 节点作业,其中主节点将协调进程并进行训练,我们将指定七个工作节点和一个主节点(这是下一个表单)。在我们的示例中,我们将使用两个总节点进行训练。因此,在表单中选择一个工作节点。
对于计算资源,我们希望在具有所有可用 GPU 资源的完整 DGX 节点上进行训练。在您的项目中选择 GPU 容量为 8 的计算资源。
在 Data Sources(数据源)窗格中,选择在上一节中创建的 PVC 的名称。本例中 PVC 的名称为
esm2-workspace。单击 Continue(继续)以导航到主节点配置页面。如前所述,对于分布式 PyTorch 作业,主 pod 可以具有与工作 pod 不同的配置。对于我们的示例,我们希望对工作节点和主节点使用相同的设置,因此请确保取消选择 Allow different setup for the master(允许主节点使用不同的设置)单选按钮。
完成后,单击 Create Training(创建训练)以将训练作业排队。
创建训练工作负载后,作业将使用 Run:ai 进行调度,并在资源可用后启动。作业将在提交后显示在 Run:ai Workloads(工作负载)页面中。完成后,模型检查点和结果将存储在您的 PVC 中,以供将来在其他下游任务(如微调或推理)中使用。有关更多信息,请参阅ESM-2nv 模型概述。