入门#
先决条件#
安装 Docker
通过运行以下命令验证您的容器运行时是否支持 NVIDIA GPU
docker run --rm --runtime=nvidia --gpus=all ubuntu nvidia-smi
示例输出
+-----------------------------------------------------------------------------+ | NVIDIA-SMI 525.78.01 Driver Version: 525.78.01 CUDA Version: 12.0 | |-------------------------------+----------------------+----------------------+ | GPU Name Persistence-M| Bus-Id Disp.A | Volatile Uncorr. ECC | | Fan Temp Perf Pwr:Usage/Cap| Memory-Usage | GPU-Util Compute M. | | | | MIG M. | |===============================+======================+======================| | 0 NVIDIA GeForce ... Off | 00000000:01:00.0 Off | N/A | | 41% 30C P8 1W / 260W | 2244MiB / 11264MiB | 0% Default | | | | N/A | +-------------------------------+----------------------+----------------------+ +-----------------------------------------------------------------------------+ | Processes: | | GPU GI CI PID Type Process name GPU Memory | | ID ID Usage | |=============================================================================| +-----------------------------------------------------------------------------+
注意
有关枚举多 GPU 系统的更多信息,请参阅 NVIDIA Container Toolkit 的 GPU 枚举文档
NGC (NVIDIA GPU Cloud) 帐户#
使用您的 NGC API 密钥进行 Docker 登录,使用
docker login nvcr.io --username='$oauthtoken' --password=${NGC_CLI_API_KEY}
NGC CLI 工具#
为您的操作系统下载 NGC CLI 工具。
重要提示
使用 NGC CLI 版本
3.41.1或更高版本。以下是在您的主目录中的 AMD64 Linux 上安装它的命令
wget --content-disposition https://api.ngc.nvidia.com/v2/resources/nvidia/ngc-apps/ngc_cli/versions/3.41.3/files/ngccli_linux.zip -O ~/ngccli_linux.zip && \ unzip ~/ngccli_linux.zip -d ~/ngc && \ chmod u+x ~/ngc/ngc-cli/ngc && \ echo "export PATH=\"\$PATH:~/ngc/ngc-cli\"" >> ~/.bash_profile && source ~/.bash_profile
在本地设置您的 NGC CLI 工具(您将需要您的 API 密钥!)
ngc config set
注意
输入您的 API 密钥后,您可能会看到组织和团队的多个选项。根据需要选择,或按 Enter 键接受默认值。
模型特定要求#
查看支持矩阵以确保您拥有受支持的硬件和软件堆栈。
启动 GenMol NIM#
拉取 NIM 容器。
docker pull nvcr.io/nim/nvidia/genmol:1.0.0
运行容器。
注意
环境变量
NGC_API_KEY必须在您的本地环境中定义且有效,以确保以下命令能够继续执行。有关个人 API 密钥设置的信息,请参阅此网站。
docker run --rm -it --name genmol-nim \ --runtime=nvidia --gpus=all -e NVIDIA_VISIBLE_DEVICES=0 \ --shm-size=2G \ --ulimit memlock=-1 \ --ulimit stack=67108864 \ -e NGC_API_KEY=$NGC_API_KEY \ -p 8000:8000 \ nvcr.io/nim/nvidia/genmol:1.0.0
打开一个新的终端并使用以下命令检查 API 的状态,直到它返回
true。这可能需要几分钟。
curl localhost:8000/v1/health/ready ... true
在 Linux Shell 中运行推理#
打开一个新的终端,保持当前终端打开并运行已启动的服务。
注意
打开一个新的终端,保持当前终端打开并运行已启动的服务。
运行推理并保存到 output.json。
curl --silent --request POST \ --url 'http://127.0.0.1:8000/generate' \ --header 'Content-Type: application/json' \ --output output.json \ --data '{ "smiles": "CCS2(=O)(=O).C134CN2C1.C3C#N.[*{15-15}]", "num_molecules": "10", "temperature": "2.0", "noise": "1.0", "step_size": "1", "scoring": "QED" }'
输出文件
output.json是 JSON 格式的内容,其中包含生成的分子 SMILES 序列和相应的分数
{
"status": "success",
"molecules":
[
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)(c2ccc(Cl)c(F)c2)C1",
"score": 0.856
},
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)(c2ccc(Cl)c(Cl)c2)C1",
"score": 0.851
},
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)(c2ccc(OC)cc2F)C1",
"score": 0.829
},
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)(c2ccc(Br)c(F)c2)C1",
"score": 0.828
},
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)(Cc2ccn(C)c2)C1",
"score": 0.812
},
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)(Cc2ccc(Br)s2)C1",
"score": 0.808
},
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)(C2CCCC(F)(F)C2)C1",
"score": 0.801
},
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)(NCCC(F)(F)F)C1",
"score": 0.789
},
...
]
}
使用 Python 运行推理#
创建一个新的空白文件,将其命名为
genmol-generate.py,并将以下内容复制到其中
import requests
import json
session = requests.Session()
response = session.post("http://127.0.0.1:8000/generate", headers = {"Accept": "application/json"}, json = {
"smiles": "CCS2(=O)(=O).C134CN2C1.C3C#N.[*{15-15}]",
"num_molecules": 5,
"temperature": 2.0,
"noise": 1.2,
"step_size":1,
"scoring": "QED"
})
response.raise_for_status()
response_body = response.json()
print(json.dumps(response_body, indent=2))
运行以下命令以启动 Python 推理脚本
python3 genmol-generate.py
下面显示了一个示例输出
{
"status": "success",
"molecules": [
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)(NC(=O)C(F)F)C1",
"score": 0.748
},
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)(C(F)F)C1",
"score": 0.728
},
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)(SC)C1",
"score": 0.717
},
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)C1",
"score": 0.634
},
{
"smiles": "CCS(=O)(=O)N1CC(CC#N)(C(=O)CC(=N)O)C1",
"score": 0.522
}
]
}
分子序列作为输入/输出#
输入是一个 SAFE 文本序列,其中包含掩码片段,作为分子生成的模板。SAFE 是 SMILES 的扩展,通过增强分子片段的表示。掩码片段是一个特殊的占位符,格式为“[*{min_len-max_len}]”,其中“min_len”和“max_len”是要为此片段生成的令牌的最小和最大数量。如果提供了空模板(仅掩码),模型将从头生成分子。如果提供了带有掩码的部分 SMILES/SAFE,模型将执行片段补全(例如,用于连接基设计、基序扩展等)。
如下图所示,前面示例中的输入分子序列从 SMILES CCS(=O)(=O)N1CC(CC#N)C1 的规范形式开始,这是 Baricitinib(一种经 FDA 批准的免疫调节药物)的基序部分。使用 safe Python 包 (链接),SMILES 文本被转换为 SAFE 格式,其中呈现了三个片段(红色、绿色和蓝色块),片段间连接点用相应原子后的成对数字(1、2 和 3)表示。然后,添加一个未配对的连接点 4 以指定装饰的位置,并在末尾添加一个作为掩码的新片段 .[*] (紫色块),它将作为生成序列提供给 GenMol 模型。
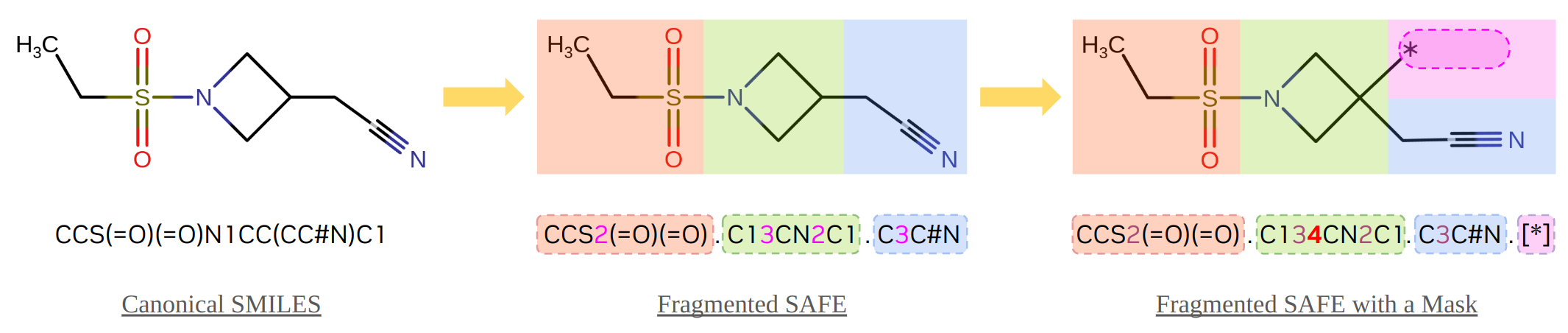
虽然模型的直接输出结果也采用 SAFE 格式,但在返回给用户之前,它们将被验证并转换回标准 SMILES 格式,以便于将结果用于其他下游工作负载。
停止容器#
当您完成端点测试后,可以通过在新的终端中运行 docker stop genmol-nim 来关闭容器。
从本地资产提供模型#
GenMol NIM 提供了实用工具,可以将模型下载到本地目录,作为模型存储库或 NIM 缓存。有关详细信息,请参阅实用工具部分。
使用之前的命令启动 NIM 容器。从那里,您可以查看和本地下载模型。
使用 list-model-profiles 命令列出可用的配置文件。
docker run -it --rm --runtime=nvidia --gpus=all -e NIM_LOG_LEVEL=WARNING \
--entrypoint list-model-profiles \
nvcr.io/nim/nvidia/genmol:1.0.0
您可以使用 download-to-cache 命令将任何配置文件下载到 NIM 缓存。例如
docker run -it --rm --runtime=nvidia --gpus=all -e NGC_API_KEY \
-v $LOCAL_NIM_CACHE:/opt/nim/.cache \
--entrypoint download-to-cache \
nvcr.io/nim/nvidia/genmol:1.0.0 \
-p a525212dd4373ace3be568a38b5d189a6fb866e007adf56e750ccd11e896b036
气隙部署(离线缓存路由)#
NIM 支持在气隙系统(也称为气墙、气隙或断开连接的网络)中提供模型。如果 NIM 检测到缓存中先前加载的配置文件,它将从缓存中提供该配置文件。在使用 download-to-cache 将配置文件下载到缓存后,可以将缓存传输到气隙系统,以运行 NIM,而无需任何互联网连接,也无需连接到 NGC 注册表。
步骤 1. 从具有互联网连接的主机下载模型,并将其保存到共享文件夹。此步骤需要在环境中使用 NGC_API_KEY。
export SHARED_NIM_CACHE=/shared/cache/folder
sudo mkdir -p $SHARED_NIM_CACHE
sudo chmod 777 $SHARED_NIM_CACHE
docker run -it --rm --runtime=nvidia --gpus=all -e NGC_API_KEY \
-v $SHARED_NIM_CACHE:/opt/nim/.cache \
--entrypoint download-to-cache \
nvcr.io/nim/nvidia/genmol:1.0.0 \
-p a525212dd4373ace3be568a38b5d189a6fb866e007adf56e750ccd11e896b036
步骤 2. 从没有互联网连接但可以访问共享文件夹的主机启动 NIM。请勿提供 NGC_API_KEY,如下例所示。
export SHARED_NIM_CACHE=/shared/cache/folder
docker run -it --rm --runtime=nvidia --gpus=all -p 8000:8000 \
-v $SHARED_NIM_CACHE:/opt/nim/.cache nvcr.io/nim/nvidia/genmol:1.0.0
气隙部署(本地模型目录路由)#
气隙路由的另一个选项是使用 NIM 容器中的 create-model-store 命令部署创建的模型存储库,以创建单个模型的存储库,如下例所示。
步骤 1. 在具有互联网连接的主机上的共享文件夹中下载并构建模型存储。此步骤需要在环境中使用 NGC_API_KEY。
export SHARED_MODEL_STORE=/shared/store/folder
sudo mkdir -p $SHARED_MODEL_STORE
sudo chmod 777 $SHARED_MODEL_STORE
docker run -it --rm --runtime=nvidia --gpus=all -e NGC_API_KEY \
-v $SHARED_MODEL_STORE:/model-store \
--entrypoint create-model-store \
nvcr.io/nim/nvidia/genmol:1.0.0 \
-p a525212dd4373ace3be568a38b5d189a6fb866e007adf56e750ccd11e896b036 -m /model-store
步骤 2. 从没有互联网连接但可以访问共享存储文件夹的主机启动 NIM。请勿提供 NGC_API_KEY,如下例所示。
export SHARED_MODEL_STORE=/shared/store/folder
docker run -it --rm --runtime=nvidia --gpus=all -p 8000:8000 \
-v $SHARED_MODEL_STORE:/model-store \
-e NIM_CACHE_PATH=/model-store \
-e MODEL_FOLDER=/model-store/genmol \
-e NIM_DISABLE_MODEL_DOWNLOAD=True \
nvcr.io/nim/nvidia/genmol:1.0.0